sep
26
 Considerada la nueva epidemia del siglo XXI, la enfermedad de Alzheimer la podrían padecer 131,5 millones de personas para 2050, de ahí la necesidad de reflexionar hoy en su Día Mundial sobre cómo prevenir este padecimiento.
Considerada la nueva epidemia del siglo XXI, la enfermedad de Alzheimer la podrían padecer 131,5 millones de personas para 2050, de ahí la necesidad de reflexionar hoy en su Día Mundial sobre cómo prevenir este padecimiento.
La Organización Mundial de la Salud (OMS) con el auspicio de Alzheimer’s Disease International (ADI), proclamó el 21 de septiembre como Día Mundial del Alzheimer, que afecta, no solo a los pacientes sino también a los cuidadores directos y familiares.
Por el momento no hay una cura para el Alzheimer, pero si se pueden realizar rutinas y actividades para retrasar el deterioro que causa la enfermedad, insisten las autoridades sanitarias.
El alzheimer es una enfermedad mental incurable que va degenerando las células nerviosas del cerebro y disminuyendo la masa cerebral, de forma que los pacientes muestran un deterioro cognitivo importante que se manifiesta en dificultades en el lenguaje, pérdida del sentido de la orientación y dificultades para la resolución de problemas sencillos de la vida cotidiana.
No existe un tratamiento para prevenir la enfermedad ni para frenar el avance. Sí que se prescriben medicamentos que ayudan con algunos síntomas de la enfermedad y para mejorar la calidad de vida de los pacientes, pero solo suele ser útil en las primeras fases, perdiendo su utilidad en fases más avanzadas.
Una vez que aparecen los primeros síntomas, los pacientes van degenerando y se van volviendo más dependientes, de forma que hay que ayudarles para vestirse, asearse, comer. Poco a poco se harán imprescindibles los cuidados permanentes de un cuidador, destacan campañas de prevención.
Se sabe que las primeras lesiones pueden aparecer unos 15 o 20 años antes de que se muestre algún síntoma, y aparecen en la región del cerebro llamada hipocampo, que se encarga, entre otras cosas, del aprendizaje y la formación de nuevas memorias. De ahí se extienden a otras partes del cerebro, cuando ya aparece el deterioro cognitivo, que finalmente desemboca en demencia.
Datos de la Organización Panamericana de la Salud (OPS) describen que la enfermedad de Alzheimer y los trastornos relacionados afectan aproximadamente a 10,3 millones de personas en las Américas y se encuentran entre las principales causas de muerte en individuos de 60 años o más.
Lamentablemente, las personas que viven con estas enfermedades son a menudo objeto de estigmatización y discriminación, advierten.
Debemos reconocer la demencia como una prioridad de salud pública e invertir en consecuencia en la reducción de riesgos, la atención en salud y asistencia social continua y las iniciativas favorables a la demencia, que promuevan la participación, la seguridad y la inclusión de las personas con demencia y sus cuidadores, reflexionó en la jornada del 2024 el director de la OPS, Dr. Jarbas Barbosa.
Una reciente publicación de la revista Lancet mostró que el 45% de todas las demencias podrían retrasarse, ralentizarse o incluso prevenirse. “Los nuevos tratamientos representan un rayo de esperanza, pero también debemos cambiar la percepción de la demencia”, subrayó Paola Barbarino, Directora General de ADI.
Esto es válido tanto para los profesionales sanitarios como para el público en general. Muchos siguen creyendo erróneamente que la demencia es una parte normal del envejecimiento, lo que niega a las personas el acceso a un diagnóstico, tratamiento, atención y apoyo oportunos, resaltó.
Para hacer frente a la carga mundial de demencia, la OMS elaboró el Plan de Acción Mundial sobre la Respuesta de Salud Pública a la Demencia 2017-2025, que fue adoptado formalmente por todos los Estados Miembros en 2017 en la 70ª Asamblea Mundial de la Salud.
El Plan describe siete áreas de acción para reducir la carga de la demencia, incluyendo el aumento del apoyo a los cuidadores de personas con demencia y la lucha contra los factores de riesgo relacionados con la demencia, como la inactividad física, la obesidad y las dietas poco saludables, entre los más significativos.
21 septiembre 2025 | Fuente: Prensa Latina | Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2025. Agencia Informativa Latinoamericana Prensa Latina S.A. | Noticia
sep
26
 Un proyecto en marcha hoy entre la Corporación FPT y el Hospital Universitario de Medicina de Hanói busca equipar a las comunidades de todo el país con desfibriladores automáticos (DEA) para mejorar la atención inicial de emergencia.
Un proyecto en marcha hoy entre la Corporación FPT y el Hospital Universitario de Medicina de Hanói busca equipar a las comunidades de todo el país con desfibriladores automáticos (DEA) para mejorar la atención inicial de emergencia.
De acuerdo con el director de la institución médica, el profesor asociado Nguyen Lan Hieu, la idea de dotar con este equipamiento a la comunidad estuvo en su mente durante muchos años, después de apreciar la popularidad de este dispositivo en lugares públicos en países desarrollados.
En Vietnam cada año mueren alrededor de 200 000 personas por enfermedades cardiovasculares, lo que representa aproximadamente un tercio del número total de fallecidos en toda la nación.
Según datos de encuestas citados por el diario Hanoi Moi, la tasa de mortalidad en pacientes con paro cardíaco extrahospitalario es muy alta, pues llega al 96,7%, mientras solo un 8,7% de los pacientes reciben reanimación cardiopulmonar en el lugar del accidente.
Respecto a la importancia de equipar a las comunidades con los DEA, la propia publicación aludió a un estudio publicado en el New England Journal of Medicine el cual reveló que las tasas de supervivencia alcanzan hasta el 74% si los pacientes recibían una descarga eléctrica en los primeros tres minutos tras un paro cardíaco.
El desfibrilador externo automático es un generador de impulsos eléctricos compacto que puede suprimir ciertas arritmias en caso de sufrir un infarto repentino.
21 septiembre 2025 | Fuente: Prensa Latina | Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2025. Agencia Informativa Latinoamericana Prensa Latina S.A. | Noticia
sep
26
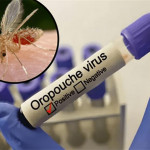 El Ministerio de Salud Pública y Población de Haití está hoy en alerta sanitaria con medidas preventivas reforzadas para evitar la expansión del oropouche en la nación caribeña.
El Ministerio de Salud Pública y Población de Haití está hoy en alerta sanitaria con medidas preventivas reforzadas para evitar la expansión del oropouche en la nación caribeña.
Según un informe preliminar, publicado por la Dirección de Epidemiología, Legislación e Investigación y el Laboratorio Nacional de Salud Pública, los primeros casos son niños varones en las edades comprendidas de cinco a 22 meses.
Estos fueron confirmados por pruebas de reacción en cadena de la polimerasa, una técnica de diagnóstico precisa que permite detectar material genético de patógenos y células anormales en muestras biológicas.
Según el sitio digital Noticias Kominotek, el 12 de septiembre fueron notificados 11 nuevos casos, que afectaron no solo a niños sino también a adultos.
Las instituciones científicas investigan activamente para evaluar el alcance de la circulación del virus y recomendar las medidas adecuadas.
Estas indicaron medidas de protección individual que incluyen el uso de repelentes, vestirse con ropa protectora, dormir con mosquiteros y la eliminación del agua limpia estancada.
De acuerdo con la literatura médica, «el virus Oropouche, es un arbovirus transmitido por mosquitos llamados bigay, y causa síntomas similares a los del dengue, chikungunya o malaria, como fiebre, escalofríos, dolor de cabeza, dolor muscular y articular».
Los científicos la consideran una dolencia leve, pero podría complicar al paciente con problemas neurológicos o hemorrágicos. Actualmente, no existe un tratamiento específico para esta enfermedad.
Actualmente, el virus Oropouche circula en varios países del Caribe, América Central y América Latina.
Precisa Noticias Kominotek, que recientemente fueron detectados casos en Guyana, Colombia, Perú, Panamá y Brasil.
20 septiembre 2025 | Fuente: Prensa Latina | Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2025. Agencia Informativa Latinoamericana Prensa Latina S.A. | Noticia
sep
26
 Cabecear repetidamente un balón de fútbol puede afectar negativamente al cerebro, incluso en jugadores aficionados que no reportan conmociones cerebrales, mostró un estudio realizado en la Universidad de Columbia, Estados Unidos.
Cabecear repetidamente un balón de fútbol puede afectar negativamente al cerebro, incluso en jugadores aficionados que no reportan conmociones cerebrales, mostró un estudio realizado en la Universidad de Columbia, Estados Unidos.
La investigación, publicada en la revista JAMA Network Open, reveló que, tras chequear a 352 futbolistas aficionados adultos, conocieron que aquellos que recibían más de mil cabezazos al año mostraron cambios microscópicos en la estructura externa del cerebro, justo detrás de los ojos, independientemente de su edad o sexo.
Hallaron además que estos jugadores también obtuvieron un rendimiento ligeramente, pero significativamente peor, en pruebas de memoria y aprendizaje.
«Lo importante de nuestro estudio es que demuestra, por primera vez, que la exposición a impactos repetidos en la cabeza provoca cambios específicos en el cerebro que, a su vez, deterioran la función cognitiva», explicó el neurocientífico Michael Lipton del mencionado centro universitario.
Los deportes de contacto, como el fútbol americano, el fútbol australiano y el rugby, también se enfrentan a las desventajas de los traumatismos craneoencefálicos repetitivos, pero en estos casos, el debate suele enmarcarse como una crisis de conmoción cerebral, generalmente reservada para los profesionales.
El estudio sugiere que incluso los golpes leves en la cabeza pueden acumularse, y no solo los atletas profesionales o quienes reportan conmociones cerebrales se ven afectados.
19 septiembre 2025 | Fuente: Prensa Latina | Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2025. Agencia Informativa Latinoamericana Prensa Latina S.A. | Noticia
sep
25
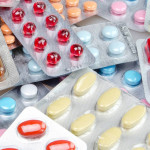 La Unión Europea (UE) registra niveles sin precedentes de desabastecimiento de fármacos, con carencias críticas en 136 medicamentos entre enero de 2022 y octubre de 2024, informó hoy el Tribunal de Cuentas Europeo.
La Unión Europea (UE) registra niveles sin precedentes de desabastecimiento de fármacos, con carencias críticas en 136 medicamentos entre enero de 2022 y octubre de 2024, informó hoy el Tribunal de Cuentas Europeo.
El documento advierte que el bloque carece de un sistema eficaz para gestionar estas crisis, mientras los problemas estructurales persisten pese a las medidas aprobadas en fecha reciente.
La situación mantiene en riesgo el acceso a tratamientos vitales, incluidos antibióticos de uso común, para la ciudadanía europea, precisan los auditores.
Analistas coinciden en que la Agencia Europea de Medicamentos vio aumentar su protagonismo durante la pandemia de covid-19.
Sin embargo, aún carece de competencias legales para auxiliar a los Estados miembros fuera de contextos de crisis sanitaria.
La creciente escasez impulsó a varias naciones a acumular existencias, una práctica que agrava el desequilibrio en el suministro por la falta de coordinación, destaca el reporte.
Por su parte, el mercado farmacéutico comunitario está fragmentado, lo cual obstaculiza la libre circulación y el aprovisionamiento adecuado de productos.
La mayoría de los medicamentos cuenta con autorización a nivel nacional, y hasta los avalados para toda la Unión Europea no se encuentran disponibles de forma homogénea, amplía la fuente.
Expertos consideran que esta falta de armonización profundiza la crisis e impide una solución colectiva efectiva al problema.
19 septiembre 2025 | Fuente: Prensa Latina | Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2025. Agencia Informativa Latinoamericana Prensa Latina S.A. | Noticia
sep
25
 La mayor biofábrica del mundo de cría de mosquitos infectados con la bacteria Wolbachia, un método que se usa para combatir el dengue, espera proteger a unos 140 millones de personas de la enfermedad en Brasil en los próximos años, informó la empresa a cargo.
La mayor biofábrica del mundo de cría de mosquitos infectados con la bacteria Wolbachia, un método que se usa para combatir el dengue, espera proteger a unos 140 millones de personas de la enfermedad en Brasil en los próximos años, informó la empresa a cargo.
La planta de Wolbito do Brasil, respaldada y usada exclusivamente por el Ministerio de Salud de Brasil, se inauguró en la ciudad de Curitiba el 19 de julio. Se trata de una empresa conjunta del World Mosquito Program, la Fundación Oswaldo Cruz y el Instituto de Biología Molecular de Paraná, que puede producir 100 millones de huevos de mosquito a la semana.
«Wolbito do Brasil podrá proteger a unos siete millones de personas en Brasil cada seis meses», dijo en una entrevista Luciano Moreira, director general de la empresa.
El dengue, conocido coloquialmente como fiebre rompehuesos por el dolor debilitante que puede causar, lo transmiten los mosquitos Aedes aegypti, que infectan a cientos de millones de personas cada año, según la Organización Mundial de la Salud (OMS).
Los casos graves de dengue pueden ser mortales, y el año pasado murieron 6 297 personas a causa de la enfermedad en Brasil, el peor año en los registros, según datos de la OMS.
La bacteria Wolbachia impide que los mosquitos transmitan el dengue y otras enfermedades como el Zika o el Chikungunya, por lo que los funcionarios de salud pública liberan mosquitos criados en laboratorio e infectados con Wolbachia para que se reproduzcan con las poblaciones locales de mosquitos y transmitan la bacteria que bloquea la transmisión del virus.
El método ya ha protegido a más de cinco millones de personas en ocho ciudades brasileñas desde 2014, según el Ministerio de Salud de Brasil.
Con la puesta en marcha de Wolbito do Brasil, los vehículos cargados de mosquitos infectados pasarán por los focos de dengue y liberarán a los insectos pulsando un botón.
«La zona elegida dentro del municipio se basa en los casos de dengue, de modo que los barrios con mayor incidencia de personas que contraen dengue son los barrios prioritarios», explica la coordinadora regional de operaciones de Wolbito do Brasil, Tamila Kleine.
19 septiembre 2025 | Fuente: Reuters | Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2025. Agencia Informativa Latinoamericana Prensa Latina S.A. | Noticia

