may
12
El 14 de abril de 2003 es una fecha importante para la historia de la ciencia. Aquel día de hace poco más de 20 años se anunció el fin del Proyecto Genoma Humano: la secuencia esencial de nuestro ADN había conseguido descifrarse después de muchos años de esfuerzo.
No obstante, ese ‘mapa’, que costó 3.000 millones de dólares y que comenzó a usarse como referencia del genoma humano, no estaba completo. Tenía lagunas en varias regiones genéticas y se basaba principalmente en el ADN de unos pocos individuos de origen europeo. Y aunque en estas dos décadas se han producido avances tecnológicos que han permitido ‘cartografiar’ esas lagunas -la secuencia completa se obtuvo en 2022- y abaratar el proceso, seguía faltando una referencia que fuera más global y diversa.
A partir de ahora, gracias a un consorcio internacional financiado por el Instituto de Investigación Nacional del Genoma Humano de EEUU, ese recurso -un pangenoma de referencia- estará disponible.
El nuevo ‘mapa’, que se presenta como un primer borrador, incluye la secuencia genética completa de 47 individuos de diferentes orígenes, lo que proporciona información detallada de 94 genomas debido a que cada individuo lleva en su ADN dos copias de genes ‘heredados’ de su padre y su madre. El objetivo del proyecto es seguir añadiendo datos al ‘mapa’, por lo que prevé que a mediados de 2024 incluya información genética de 350 personas de ascendencia étnica diversa.
«Hasta ahora, la referencia que usaba la comunidad científica estaba incompleta y carecía de diversidad», señaló en rueda de prensa Benedict Paten, director asociado del Instituto de Genómica Santa Cruz de la Universidad de California y uno de los líderes del proyecto. Este nuevo recurso, en cambio, proporciona una imagen más completa y permitirá realizar análisis más certeros a la hora de caracterizar la variabilidad genética de la población humana sea cual sea su origen, destacó.
De hecho, el nuevo pangenoma ya ha sacado a la luz más de 100 millones de nuevas bases -cada una de las letras que componen el genoma-, y ha destapado nuevos alelos en regiones estructuralmente complejas del genoma que hasta ahora no figuraban en el genoma de referencia. Los detalles de la investigación se publican en cuatro artículos en el último número de las revistas Nature y Nature Biotechnology.
Mediante técnicas computacionales de última generación, los investigadores han sido capaces de construir un recurso que, en lugar de ser único y lineal, como era hasta ahora la referencia GRCh38 que se utilizaba, aporta distintas versiones de una misma secuencia al mismo tiempo, lo que proporciona a los investigadores un mayor abanico de opciones para sus análisis. En el proyecto ha participado un equipo del Centro de Supercomputación de Barcelona liderado por Santiago Marco-Sola.
Qué supone un nuevo pangenoma para la investigación
«Hasta ahora nos hemos contentado con una sola secuencia del genoma que en su día se decidió arbitrariamente que era la secuencia referencia, formada por trozos de secuencia de un puñado de personas de ascendencia principalmente europea. Y si bien esto ha tenido una gran utilidad, también tiene muchas limitaciones», señala Jorge Ferrer, investigador del Centro de Regulación Genómica de Barcelona (CRG). «Por ejemplo, aunque resulte sorprendente, a cada uno de nosotros nos pueden faltar o sobrar unos cuantos trozos muy grandes del genoma. Si el pedazo de genoma escogido para ser la referencia es de alguien que no tiene ese trozo (o lo tiene suficientemente alterado), el mapa de referencia que utilizamos actualmente no serviría para una persona que tiene una mutación que afecta esa parte», aclara. Para complicar aún más las cosas, continúa, «el genoma puede variar enormemente en diferentes partes del mundo. Y si el mapa de referencia está hecho con variantes europeas, tiene menor utilidad para interpretar el genoma de una persona de Camerún o China».
El trabajo actual, apunta Ferrer, «es el primer paso para resolver estos problemas». «Han creado un sistema complejo que permite cotejar la secuencia genómica de una persona con todas estas posibles secuencias humanas, en lugar de con una sola secuencia y el consorcio tiene planes de desarrollar esta estrategia con la secuencia de muchos más individuos».
Para José Manuel Castro Tubío, líder del Grupo de Investigación de Genomas y Enfermedad del Centro de Investigación en Medicina Molecular y Enfermedades Crónicas (CIMUS) de Santiago, este nuevo recurso va a ayudar, en primer lugar, a «conocer mejor nuestra identidad, a conocer qué es lo que nos hace diferentes genéticamente a unos de otros». Y el hecho de «conocer lo que nos hace diferentes, qué secuencias de material genético nos hacen diferentes, nos va a permitir saber cosas acerca de nuestra evolución y nos va a permitir también saber cosas sobre las enfermedades genéticas que nos afectan».
«La variabilidad genética va asociada a rasgos biológicos y también a la predisposición de desarrollar enfermedades», explica. «Estos nuevos genomas que ahora se publican van a permitir descubrir muchas variantes que todavía no sabemos a qué se asocian».
Si bien la nueva referencia presentada todavía es un primer borrador y solo representa a un número todavía pequeño de individuos, contiene información que va a ser muy útil para avanzar en la investigación biomédica, concluye el investigador.
Mayo 10/2023 (Diario Médico) – Tomado de Genética – Investigación Copyright Junio 2018 Unidad Editorial Revistas, S.L.U. Todos los derechos reservados
may
11
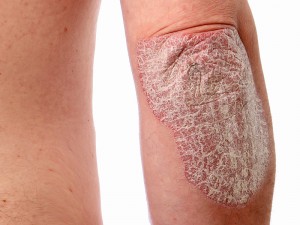
Trece hospitales de la Comunidad Valenciana están inmersos en un estudio multidisciplinar que pretende evaluar la adherencia, eficacia y persistencia de los tratamientos biológicos prescritos a pacientes con psoriasis moderada-grave en seguimiento durante un año. Para ello, los tres investigadores principales, Raul Ferrando, jefe del Servicio de Farmacia del Hospital General Universitario de Castellón; Antonio Martorell, jefe de Dermatología del Hospital de Manises, de Valencia, y Joaquín Borrás, farmacéutico de la Unidad de Pacientes Externos del Hospital de Sagunt, de Valencia, pensaron que, para facilitar el registro de datos clínicos, farmacoterapéuticos y de calidad de vida era necesario crear una app. Pero no una aplicación cualquiera sino una que permitiera que el farmacéutico, el dermatólogo y el paciente pudieran introducir datos, cada uno en su campo. Y así lo hicieron y la han llamado PsorUp, ya disponible en IOS y Android, y es de registro solo para los investigadores y pacientes incluidos en el ensayo.
Para aclarar conceptos, Ferrando recuerda a CF que la adherencia es cuando «el paciente se toma la medicación de forma adecuada». Esto está directamente asociado a la eficacia, pues «hay una evidencia clara de que, si un paciente es adherente, la eficacia del tratamiento es mayor y, como consecuencia de esa eficacia, el tratamiento se mantiene en el tiempo sin cambios, que es la persistencia», explica.
«Esta trilogía era lo que pretendíamos valorar en los tratamientos biológicos de la psoriasis moderada-grave -prosigue- y pensamos que eso se podía gestar de una manera más fácil con la creación de una app, que nos permitiera obtener esos datos e implicar a todos los participantes en este acto sanitario, que son el dermatólogo, el farmacéutico hospitalario y el paciente».
Así, el dermatólogo introduce todos los datos clínicos del paciente y el seguimiento de los resultados de salud a través de las escalas de psoriasis. «Hablamos de datos relacionados con el diagnóstico de la enfermedad, qué tratamientos ha llevado, qué es lo que se prescribe, cómo se encuentra el paciente, cuáles son sus escalas de valoración clínica, si el paciente va mejor o peor… Y son datos que actualiza en las sucesivas visitas que tenga con el paciente».
El farmacéutico, por su parte, registra los tratamientos que se le da y dónde se administra, «ya que puede ser en el hospital, en consulta o se la puede administrar el propio paciente…», aclara. La fecha de recogida y cómo será la recogida, entre otros datos relacionados con la adherencia, haciendo test de Morisky Green. Además, se le pregunta si se le olvida alguna vez o si tiene algún problema.
El paciente también cuenta en esta app, pues, al inicio y en cada una de las visitas siguientes, va introduciendo datos sobre cómo se encuentra, siguiendo escalas de calidad de vida. «Es lo que se conoce como PRO (resultados en salud percibidos por el paciente). Le preguntamos por su calidad de vida, si la psoriasis le está afectando en su día a día, si tiene molestias o picor… Todo vinculado a su percepción, no a la del médico, que él ya pone la suya. Y es que, el clínico puede ver que no hay una afectación cutánea en el cuerpo, pero el paciente puede percibir que no está bien, ya sea emocionalmente o físicamente, y esto hay que alinearlo para ver si los resultados del tratamiento están siendo óptimos».
Según Ferrando, «la potencia de la app radica en la suma de toda esta información» y lo que la diferencia respecto a otras es que «la van a tocar todos: el dermatólogo, el farmacéutico y el paciente, que la van a nutrir de información». A lo que Martorell añade: «A día de hoy no existe una aplicación como el modelo PsorUp, que ponga al paciente en el centro del proceso, lo que permitirá incrementar su proactividad, dándole el papel protagonista que siempre debe de tener». Es más, asegura que «PsorUp es la primera app incluida en las tiendas de IOS y Google a nivel global enfocada a mejorar conocimientos de la adherencia terapéutica de los pacientes con psoriasis incluidos en un proyecto de investigación». Asimismo, califica de «hito colaborativo» que el contenido de la app se haya preparado de forma conjunta por dermatólogos y farmacéuticos hospitalarios de varios centros de la Comunidad Valenciana.
El farmacéutico especialista concreta que los investigadores harán un seguimiento de un año, en función de las visitas programadas por el dermatólogo. En cada una de ellas se hará el registro de datos por parte de los implicados.
Al final de la investigación, verán si la app ha servido para recoger todos los datos y si les ha permitido hacer un seguimiento de la adherencia, la persistencia y la evaluación de los resultados en salud.
Mayo 11/2023 (Diario Médico) – Tomado de Farmacia Hospitalaria – En investigación Copyright Junio 2018 Unidad Editorial Revistas, S.L.U. Todos los derechos reservados.
may
10
Investigadores de la Facultad de Artes y Ciencias de la Universidad de Pensilvania y de la Queen’s University de Canadá han elaborado un modelo teórico de transmisión de la enfermedad Covid-19 que tiene en cuenta los efectos de la dinámica social y, en concreto, el modo en que las normas sociales afectan a las Intervenciones No Farmacéuticas (INF), como el enmascaramiento y el distanciamiento social.
La investigación, publicada en Proceedings of the National Academy of Sciences, muestra que la conformidad social crea una especie de «adherencia» por la que los individuos se niegan a cambiar su uso de las INF si difiere de lo que hacen los demás.
Los investigadores pretendían comprender mejor cómo la priorización del riesgo y las normas sociales afectan a la adopción de las INF durante una pandemia. Para ello, desarrollaron un modelo que tiene en cuenta el riesgo de infección, el coste de las INF y el coste social de desviarse de las normas de uso de las INF. El modelo describe la dinámica de umbral en el número de individuos necesarios para apoyar un cambio de comportamiento, lo que crea «puntos de inflexión» en la adopción de comportamientos INF donde un pequeño cambio en la prevalencia de la enfermedad puede causar un cambio significativo en el comportamiento de la población.
«Nuestro modelo descubrió que pequeños cambios en determinados factores, como la eficacia de las INF, la tasa de transmisión y los costes de las intervenciones, pueden provocar grandes cambios en la tasa de propagación de la enfermedad, o tasa de ataques», afirma Akçay.
Explica que esto se debe en parte a que la gente es conformista y, por tanto, lenta a la hora de adoptar nuevos comportamientos como el uso de mascarillas, hasta que la enfermedad alcanza niveles tan altos que la percepción del riesgo anula la conformidad, momento en que la población se vuelca. El conformismo también funciona en sentido contrario; el nuevo comportamiento persiste más tiempo en la población de lo que lo haría si la gente sólo se preocupara de sus riesgos y costes individuales. Esto crea olas de infección y de comportamiento ante INF distintas.
Según el equipo, los resultados ponen de manifiesto una compleja relación entre las normas sociales y la propagación de la enfermedad. Explican que al realizar una simulación epidemiológica sin uso de INF al principio de la epidemia, obtuvieron una tasa de ataques previsiblemente alta y, con el tiempo, los individuos empezaron a utilizar INF por temor al riesgo de infección.
Sin embargo, el inicio del uso del INF se produce mucho más tarde, cuando los parámetros del coste de desviarse de las normas sociales se fijan más altos «porque si nadie se enmascara no quieres ser la primera persona», afirma Akçay.
«Así que aumentar este parámetro provoca un retraso en el enmascaramiento, lo que hace que la primera oleada de la epidemia sea mucho más alta de lo que habría sido si los individuos reaccionaran a sus niveles de riesgo. Por otra parte, cuando realizamos la simulación con enmascaramiento y el número de casos empezó a descender, se produjo una reticencia a dejar de enmascararse porque nadie quería ser la primera persona en dejar de hacerlo, lo que denominamos pegajosidad.»
Morsky explica que el modelo estuvo motivado inicialmente por algunos resultados de un estudio anterior que investigaba las normas sociales y sus efectos en relación con el comportamiento de reciprocidad, donde el comportamiento conformista puede inducir ciclos de auge y caída en comunidades cooperativas. En este caso, el comportamiento conformista hace que las oleadas epidémicas sean intrínsecamente más distintas de lo que habrían sido de otro modo, inclusive en ausencia de factores externos como la variación estacional de las tasas de transmisión.
Akçay afirma que la información sobre estas tendencias y dinámicas sociales puede ser útil para los responsables políticos que sopesan decisiones sobre cómo responder al comportamiento humano.
Mayo 10/2023 (MedicalXpress) – Tomado de Diseases, Conditions, Syndromes – Health informatics Copyright Medical Xpress 2011 – 2023 powered by Science X Network.
Traducción realizada con la versión gratuita del traductor www.DeepL.com/Translator
may
10
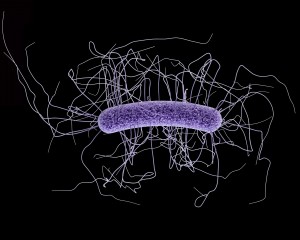
Un equipo de bioquímicos de la Universidad de Notre Dame ha hallado una posible nueva forma de tratar a los pacientes infectados por Clostridium difficile, un tipo de bacteria que causa graves problemas gastrointestinales. En su estudio, publicado en Proceedings of the National Academy of Sciences, el grupo buscó en bases de datos médicas moléculas antibacterianas que pudieran funcionar mejor en pacientes con infecciones por C. difficile.
Las infecciones por C. difficile pueden provocar desde pequeñas molestias hasta episodios potencialmente mortales: cada año mueren unas 13.000 personas. Una vez infectados (muy a menudo mientras están hospitalizados por otra causa), los pacientes suelen recibir un antibiótico para tratar la infección. Desgraciadamente, los antibióticos son cada vez menos eficaces a medida que C. difficile desarrolla resistencias.
Actualmente sólo se utiliza uno: la vancomicina. Otro problema es que C. difficile produce esporas que no son eliminadas por el antibiótico, lo que significa que muchas personas sufren reinfecciones. La vancomicina no funciona bien en estos pacientes, y menos aún en infecciones posteriores. Por estas razones, el equipo de Indiana se propuso encontrar un tratamiento más eficaz para estas infecciones.
El trabajo consistió en buscar en bases de datos antibacterianas moléculas que se sabe que se unen a una proteína específica asociada a C. difficile. Así se llegó a dos compuestos: los oxadiazoles 1 y 2. Las pruebas in vitro demostraron que ambos eran eficaces para eliminar la C. difficile cuando se administraban a la misma dosis que la vancomicina.
A continuación, el equipo comprobó la rapidez con la que se absorbían en el torrente sanguíneo si se administraban por vía oral. El oxadiazol 1 se absorbía con rapidez, lo que lo hacía inadecuado para combatir las bacterias intestinales. En cambio, el oxadiazol 2 se absorbía lentamente, lo que lo convertía en el candidato ideal para una nueva terapia contra las infecciones por C. difficile. En ratones, previno la muerte un 30% mejor que la vancomicina.
El estudio también descubrió que el oxadiazol 2 ayudaba a prevenir la pérdida de peso mejor que la vancomicina. Y lo mejor de todo es que las pruebas fecales no mostraron indicios de esporas: tras tres semanas de pruebas, ninguno de los ratones presentaba reinfecciones.
Mayo 10/2023 (MedicalXpress) – Tomado de Medications – Gastroenterology Copyright Medical Xpress 2011 – 2023 powered by Science X Network.
Traducción realizada con la versión gratuita del traductor www.DeepL.com/Translator
may
3
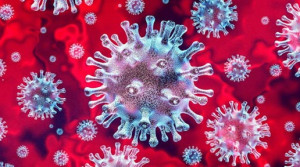
Tras un estudio dirigido por la Universidad de Queensland sobre la respuesta de las células inmunitarias al virus, se está más cerca de encontrar un tratamiento que evite la inflamación galopante en pacientes con COVID-19 grave.
La doctora Larisa Labzin y la profesora Kate Schroder, del Instituto de Biociencia Molecular de la UQ, junto con la doctora Sarah Londrigan, del Instituto Peter Doherty de Infección e Inmunidad, han descubierto que la mayoría de las células inmunitarias que contribuyen a la inflamación crónica no están infectadas por el virus SARS-CoV-2 que causa la COVID-19.
Según el Dr. Labzin, en lugar de desencadenar una respuesta protectora para eliminar el virus, estas células no infectadas llamadas macrófagos detectan daños y muerte en las células vecinas y desencadenan una fuerte respuesta inflamatoria.
«Se produce un desequilibrio en la respuesta inmunitaria porque la mayoría de los macrófagos no están infectados por el virus», explica el Dr. Labzin.
«Acabamos con demasiadas células inmunitarias que acuden al lugar de la infección y causan muchos daños colaterales: demasiada inflamación y poca lucha contra el virus».
«Es un arma de doble filo para el organismo: el sistema inmunitario que ataca una enfermedad infecciosa en una fase temprana es protector, pero cuando es prolongado o excesivo, puede provocar una inflamación crónica».
El equipo de investigación del IMB está estudiando cómo actuar selectivamente sobre los macrófagos sin comprometer la capacidad del organismo para luchar contra el virus, con el fin de reducir la incidencia de la COVID grave.
En la actualidad se administran antiinflamatorios a los pacientes hospitalizados por COVID-19 después de que el virus haya alcanzado su pico máximo, para calmar la respuesta inmunitaria hiperactiva, pero los fármacos hacen que los pacientes sean susceptibles a infecciones secundarias.
Con los nuevos conocimientos sobre el funcionamiento de los macrófagos, los investigadores pretenden diseñar antiinflamatorios que puedan administrarse antes para evitar que la inflamación se descontrole.
Según la profesora Kate Schroder, conocer mejor la biología fundamental del sistema inmunitario nos ayudará a combatir mejor las infecciones.
«Tenemos vacunas y antivirales para luchar contra el COVID-19, pero el virus sigue mutando, así que ésta es una forma de prepararnos para el futuro contra nuevas variantes y también contra futuras pandemias e infecciones».
Más información: Larisa I. Labzin et al. (2023). Macrophage ACE2 is necessary for SARS-CoV-2 replication and subsequent cytokine responses that restrict continued virion release. Science Signaling. https://www.science.org/doi/10.1126/scisignal.abq1366
Mayo 3/2023 (MedicalXpress) – Tomado de Immunology – Inflammatory disorders Copyright Medical Xpress 2011 – 2023 powered by Science X Network
Traducción realizada con la versión gratuita del traductor www.DeepL.com/Translator
may
3
Un equipo de 87 investigadores de varias instituciones dirigido por la Facultad de Salud Pública de la Universidad de Michigan ha publicado un artículo en Science Advances titulado «The genetic determinants of recurrent somatic mutations in 43,693 blood genomes» (Los determinantes genéticos de las mutaciones somáticas recurrentes en 43.693 genomas sanguíneos), que cuestiona algunos supuestos largamente sostenidos sobre las mutaciones somáticas no oncogénicas.
El grupo analizó 43.693 genomas completos de muestras de sangre de 37 cohortes e identificó 7.131 mutaciones somáticas sin sentido recurrentes en al menos 50 individuos. Según los investigadores, estas mutaciones somáticas recurrentes sin sentido (RNMSM) no se explican claramente por otros fenómenos clonales, como la hematopoyesis clonal.
Las mutaciones somáticas son ligeras variantes en los genes que se adquieren en vida después del nacimiento. Son comunes en todos los animales y suelen suponer un riesgo patológico bajo a menos que se produzcan en una proteína funcional. Las mutaciones somáticas son, por definición, mutaciones que no afectan a las células sexuales, por lo que no son heredables.
Se observó que la prevalencia de las RNMSM aumenta con la edad, como cabría esperar, ya que una persona de 50 años de media tiene 27 RNMSM. Lo que puede no esperarse es una variación hereditaria de la línea germinal asociada a la adquisición de RNMSM. Estas variantes se encontraron en genes implicados en la función inmunitaria adaptativa, la producción de citocinas proinflamatorias y el compromiso de linaje linfoide. Los investigadores también hallaron ocho RNMSM asociadas a rasgos de las células sanguíneas con tamaños de efecto comparables a los de las mutaciones genéticas mendelianas.
Los autores afirman que se cree que las mutaciones somáticas no oncogénicas son infrecuentes e intrascendentes, pues tienen poco que ver con la función celular, ya que no alteran las proteínas ni causan patologías. Sin embargo, los investigadores descubrieron que las mutaciones somáticas en la sangre son un fenómeno inesperadamente común, con determinantes específicos según la ascendencia y consecuencias para la salud humana.
Los sitios de variantes no codificantes asociados positivamente a los rasgos de las células sanguíneas son especialmente interesantes. Dado que no se cree que contribuyan a los rasgos o la patología, estos cambios han pasado desapercibidos y, por tanto, no se ha estudiado su posible relación con la manifestación de enfermedades.
Los hallazgos de los autores sobre una clase de variación genética, ampliamente presente en todo el genoma humano, con determinantes genéticos de la línea germinal y consecuencias fenotípicas refutan directamente lo que antes se suponía pero nunca se había estudiado.
Mayo 3/2023 (MedicalXpress) – Tomado de Genetics Copyright Medical Xpress 2011 – 2023 powered by Science X Network
Traducción realizada con la versión gratuita del traductor www.DeepL.com/Translator