sep
12
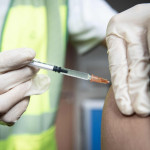 China sometió este lunes a ensayos clínicos su primera vacuna propia contra la viruela del mono. La vacuna, desarrollada de forma independiente por el Instituto de Productos Biológicos de Shanghai, adscrito al Grupo Farmacéutico Nacional de China (Sinopharm), se basa en un ortopoxvirus vivo atenuado, la Vaccinia Ankara Modificada (MVA).
China sometió este lunes a ensayos clínicos su primera vacuna propia contra la viruela del mono. La vacuna, desarrollada de forma independiente por el Instituto de Productos Biológicos de Shanghai, adscrito al Grupo Farmacéutico Nacional de China (Sinopharm), se basa en un ortopoxvirus vivo atenuado, la Vaccinia Ankara Modificada (MVA).
De acuerdo al Instituto de Productos Biológicos de Shanghai, esta cepa MVA ha demostrado su seguridad y eficacia como vector candidato. La vacuna contra la viruela del mono MVA se produce mediante un proceso de producción de células maduras que es estable y confiable en su calidad. Los estudios preclínicos han demostrado su seguridad y su capacidad para generar una protección inmunitaria eficaz contra el virus de la viruela del mono en modelos de primates no humanos.
Entre enero de 2022 y agosto de 2024, más de 120 países han notificado casos de viruela símica, con más de 100 000 casos confirmados por laboratorio y más de 220 muertes entre los casos confirmados, informó la Organización Mundial de la Salud (OMS).
Junto con la aparición y rápida propagación de una nueva cepa de virus, el 14 de agosto, la OMS declaró la viruela del mono como emergencia de salud pública de importancia internacional en la República Democrática del Congo (RDC) y los países vecinos, el nivel más alto de alerta de la OMS.
Lu Hongzhou, director del Tercer Hospital Popular de Shenzhen, considera que, aunque el brote actual se centró en la RDC, no se puede descartar la posibilidad de que la cepa 1b se haya extendido por todo el mundo.
«Sobre la base de las medidas actuales de prevención y control y el sistema nacional de monitoreo de epidemias, la probabilidad de un rápido aumento en las infecciones en China por viruela del mono sigue siendo relativamente baja», aseguró Lu, y pidió a que la población se mantenga alerta e informada.
10 septiembre 2024|Fuente: Diario del Pueblo |Tomado de |Noticia
sep
6
 Expertos en biología molecular se citan desde este martes y hasta el viernes en el Palacio de Exposiciones y Congresos de A Coruña en el 46º Congreso de la Sociedad Española de Biología y Biología Molecular (SEBBM), que abordará temas como el cáncer, para el que «falta ciencia» para superarlo, la biomedicina o la inteligencia artificial.
Expertos en biología molecular se citan desde este martes y hasta el viernes en el Palacio de Exposiciones y Congresos de A Coruña en el 46º Congreso de la Sociedad Española de Biología y Biología Molecular (SEBBM), que abordará temas como el cáncer, para el que «falta ciencia» para superarlo, la biomedicina o la inteligencia artificial.
En la presentación, la presidenta del comité organizador, María Mayán, ha puesto el foco sobre las terapias dirigidas, que «salvan vidas ya, aunque el porcentaje es del 20 %» por lo que «se necesita más investigación» y abaratar los costes de este tipo de tratamientos: «falta mucha ciencia para superar la pandemia del cáncer», resume.
Mayán ha insistido en la necesidad de «aumentar el uso de estas terapias», pero ha recordado que faltan por «identificar» ciertos aspectos del cáncer para que «los pacientes respondan bien».
La presidenta saliente de la SEBBM, Isabel Varela, ha indicado que otro de los retos es «reducir el precio de esas terapias para extenderlas a toda la población».
Varela ha asegurado que este congreso, que se celebra por segunda vez en A Coruña tras la edición de 2003, es «una gran oportunidad para la ciencia española».
La vocal de congresos de la Sociedad y organizadora de la cita del próximo año en Cáceres, Guadalupe Sabio, ha manifestado que la función de la SEBBM es «llevar la ciencia a la ciudad», es decir, «no solo nutrir a los científicos» sino «implicar a las ciudades en las que se celebran los congresos».
Por eso, esta cita cuenta también con actividades de divulgación como una exposición de Severo Ochoa en la Domus o una actividad que mezcla ciencia y música con la actuación de Fillas de Cassandra.
Otro reto, ha indicado el próximo presidente de la SEBBM, Antonio Ferrer, es «romper el mito de que la ciencia es inaccesible para la sociedad».
«Los científicos tenemos ese compromiso social, queremos dar formación a la sociedad a través de la ciencia y evitar la desinformación que es tan severa en estos momentos por las redes sociales», ha declarado, a la vez que ha apuntado que este congreso puede servir para «motivar a los jóvenes a iniciar sus carreras profesionales dentro de la biología molecular».
Este encuentro en A Coruña arranca este martes con la charla del inmunólogo Tak W. Mak y concluye el viernes, 6 de septiembre, con la intervención de Evan Rosen, quien ha desarrollado investigaciones punteras relacionadas con la obesidad.
Así, más de 700 profesionales se reunirán en este congreso para analizar los avances de la biología y la biología molecular, que abarca diferentes áreas, por lo que servirá para «inspirar y establecer colaboraciones multidisciplinares», ha resumido Mayán.
03 septiembre 2024|Fuente: EFE |Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2024. Agencia Informativa Latinoamericana Prensa Latina S.A.|Noticia
sep
5
 Un equipo de investigadores ha logrado, por primera vez, crear células madre sanguíneas muy parecidas a las del cuerpo humano y que han probado con ratones; este descubrimiento podría llevar en un futuro a tratamientos personalizados contra la leucemia y otros trastornos por insuficiencia de la médula ósea.
Un equipo de investigadores ha logrado, por primera vez, crear células madre sanguíneas muy parecidas a las del cuerpo humano y que han probado con ratones; este descubrimiento podría llevar en un futuro a tratamientos personalizados contra la leucemia y otros trastornos por insuficiencia de la médula ósea.
El estudio encabezado por el Instituto Murdoch de Investigación Infantil (MCRI) de Melbourne (Australia) y que publica Nature Biotechnology ha superado un importante obstáculo para producir células madre sanguíneas humanas, que pueden crear glóbulos rojos, glóbulos blancos y plaquetas, muy parecidas a las del embrión humano.
El avance conseguido por el equipo, encabezado por Elizabeth Ng, del MCRI, allana el camino para que estas células cultivadas en laboratorio puedan utilizarse en trasplantes de células madre sanguíneas y de médula ósea.
«La capacidad de tomar cualquier célula de un paciente, reprogramarla como célula madre y convertirla en células sanguíneas específicamente compatibles para el trasplante tendrá una enorme repercusión en la vida de estos pacientes vulnerables», dijo la investigadora en un comunicado del MCRI.
Hasta ahora, no era posible desarrollar en el laboratorio células madre sanguíneas humanas que pudieran trasplantarse a un modelo animal de médula ósea incapaz de producir células sanguíneas sanas.
«Hemos desarrollado un flujo de trabajo que ha creado células madre sanguíneas trasplantables que se asemejan mucho a las del embrión humano», que además pueden crearse a escala y con la pureza necesaria para su uso clínico, agregó.
El equipo inyectó estas células madre sanguíneas humanas manipuladas en laboratorio a ratones inmunodeficientes y vieron cómo se convertían en médula ósea funcional a niveles similares a los observados en los trasplantes de células de sangre de cordón umbilical.
La investigación también descubrió que las células madre cultivadas en laboratorio podían congelarse antes de ser trasplantadas con éxito a los ratones, lo que imitaba el proceso de conservación de las células madre de la sangre del donante antes de ser trasplantadas a los pacientes.
Estos hallazgos podrían dar lugar a nuevas opciones terapéuticas para diversos trastornos sanguíneos, según el también firmante del trabajo Ed Stanley, del MCRI.
«Los glóbulos rojos son vitales para el transporte de oxígeno y los blancos son nuestra defensa inmunitaria, mientras que las plaquetas provocan la coagulación para detener las hemorragias; entender cómo se desarrollan y funcionan estas células es como descifrar un complejo rompecabezas», explicó.
Al perfeccionar los métodos con células madre que imitan el desarrollo de las células madre sanguíneas que se encuentran en nuestro cuerpo «podemos comprender y desarrollar tratamientos personalizados para una serie de enfermedades de la sangre, incluidas las leucemias y la insuficiencia de la médula ósea».
Aunque el trasplante de células madre sanguíneas suele ser un elemento clave en el tratamiento de las hemopatías infantiles, no todos los niños encuentran un donante idóneo, recordó el también autor del estudio Andrew Elefanty.
Sin embargo, el desarrollo de células madre sanguíneas personalizadas y específicas para cada paciente evitará estas complicaciones, «abordará la escasez de donantes y, junto con la edición del genoma, ayudará a corregir las causas subyacentes de las enfermedades de la sangre», señaló.
La siguiente fase de la investigación, que probablemente se lleve a cabo en unos cinco años con financiación pública, consistirá en realizar un ensayo clínico de fase uno para comprobar la seguridad del uso en humanos de estas células sanguíneas cultivadas en laboratorio.
03 septiembre 2024|Fuente: EFE |Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2024. Agencia Informativa Latinoamericana Prensa Latina S.A.|Noticia
sep
2
 Hace 30 años que la ciencia no descubre un antibiótico nuevo, por eso, señala el investigador César de la Fuente, hay que buscar moléculas «en todas partes», incluso en nuestros parientes más cercanos, como los neandertales, y en otros organismos extintos, como el mamut: «Queremos explorar todo el árbol de la vida».
Hace 30 años que la ciencia no descubre un antibiótico nuevo, por eso, señala el investigador César de la Fuente, hay que buscar moléculas «en todas partes», incluso en nuestros parientes más cercanos, como los neandertales, y en otros organismos extintos, como el mamut: «Queremos explorar todo el árbol de la vida».
De la Fuente trabaja en la Universidad de Pensilvania, Estados Unidos, y lleva casi una década aplicando herramientas de inteligencia artificial (IA) para rebuscar, en cada rincón de la biología -viva y extinta-, moléculas con potencial antibiótico, para frenar lo que cada vez más es un problema de salud mundial: las bacterias resistentes.
«Hemos logrado acelerar dramáticamente nuestra capacidad para descubrir nuevos antibióticos», indica en entrevista telemática con EFE el investigador español; a día de hoy, forman parte de su ‘biblioteca molecular’ más de un millón de péptidos -cadenas cortas de aminoácidos conocidas por su potencial como antibióticos innovadores-.
Que él y su equipo han encontrado en neandertales, denisovanos, mamuts lanudos, elefantes de colmillos rectos y perezosos gigantes, todas ellas extintas, y en la saliva y el microbioma humano, en vísceras de cerdo, plantas y muchos otros organismos marinos y terrestres.
¿Por qué no hay nuevos antibióticos?
De la Fuente, quien ha recibido numerosos galardones, como el Premio Princesa de Girona de Investigación en 2021 o el premio Langer, explica que son múltiples los factores que han entorpecido el hallazgo de antibióticos totalmente nuevos -solo se han «modificado mínimamente» las estructuras de algunos-.
Cada vez, dice, hay menos inversión y no hay incentivos a nivel de mercado. Además, durante mucho tiempo se pensó que el problema -combatir bacterias- estaba resuelto porque existían fármacos que funcionaban, lo que «desincentivó» a científicos y compañías, que dirigieron su mirada al cáncer y otras enfermedades.
Pero con el tiempo, como ya advirtió Alexander Fleming -descubridor de la penicilina- en su discurso de aceptación del Nobel, las bacterias se han ido haciendo cada vez más resistentes: existe una brecha de muchos años sin innovación en nuevos antibióticos.
A esto, añade el investigador de A Coruña, hay que sumar que los métodos tradicionales de muestreo y laboratorio para hallar moléculas novedosas «están un poquito obsoletos. Descubrir algo interesante puede llevar entre 6 y 7 años, más tiempo del que se tarda en completar un doctorado, y ni siquiera está garantizado».
Aquí, asevera, es donde entran en juego las máquinas: «los algoritmos pueden acelerar el proceso».
Se trata de aprovechar varias décadas de información biológica disponible en forma de secuenciación de proteomas -el conjunto de proteínas producidas por un organismo y codificadas en su genoma-; genomas -todos los genes de un organismo-; y metagenomas -el conjunto completo de material genético presente en una comunidad microbiana en un entorno específico-.
Y aplicar luego los algoritmos adecuados para encontrar, en esa inmensidad de datos, moléculas escondidas. «Es hacer ciencia a velocidad digital», declara el investigador, quien subraya que siempre, para verificar que lo identificado por la IA es correcto, hay que sintetizar en el laboratorio algunos péptidos.
Además, es fundamental probar su actividad antimicrobiana ‘in vitro’ y en modelos animales, lo que De la Fuente ha logrado.
La ‘desextinción molecular’
El primer paso de esta aventura científica fue escudriñar el proteoma humano; gracias a la IA, se identificaron por primera vez miles de péptidos ocultos con potencial antibiótico. Eso hizo plantearse al equipo que quizás no solo estuvieran ahí, sino también conservados a lo largo de la evolución, del árbol de la vida.
Comenzaron a explorar moléculas similares en neandertales y denisovanos y a desarrollar un nuevo paradigma, la «desextinción molecular», que se refiere al proceso de resucitar moléculas de especies extintas para «hacer frente a problemas de hoy en día».
La primera de estas moléculas extintas reconocida -hubo que inventarse un nombre- fue la ‘neandertalina-1′; «fue emocionante», admite el científico. Después vinieron otras y la idea de ir más allá de los antepasados humanos, ampliando la búsqueda a todas las especies extintas conocidas por la ciencia.
Para lograrlo, De la Fuente y su equipo desarrollaron un nuevo modelo de aprendizaje profundo, denominado APEX, entrenado con bases de datos de ADN arcaico de diversos organismos. Este modelo se enfocó en el «extintoma», la información genética de organismos extintos.
Descubrieron la ‘mamutusina-2′ del mamut lanudo, la ‘elefasina-2′ del elefante de colmillos rectos o la ‘hidrodamina-1′ de la antigua vaca marina. Algunas moléculas mostraron en ratones una eficacia comparable a la del antibiótico polimixina B, comúnmente usado en hospitales, afirma De la Fuente, para quien aún faltan datos para saber si las moléculas extintas tienen mayor potencial que las de organismos vivos.
Cuestiones bioéticas
El equipo está pensando ahora los siguientes pasos, porque el objetivo final es desarrollar antibióticos nuevos. Una opción es crear ellos mismos una empresa (‘spin-off’) para explotar los resultados y diseñar nuevos mecanismos para implementar ensayos clínicos, quizás a través de una fundación sin ánimo de lucro. «El mercado está roto y hay que explorar nuevas vías».
La desextinción es un campo nuevo y su desarrollo ha abierto un debate ético y sobre cómo patentar estas moléculas.
«Al abordar el concepto de resucitar moléculas del pasado, nos enfrentamos a profundas cuestiones éticas sobre nuestra relación con la naturaleza, los límites de la intervención humana y nuestras responsabilidades como administradores del mundo biológico», escribió recientemente en la revista Nature Biotechnology el científico gallego junto al experto en patentes y bioética Andrew W. Torrance.
En las conversaciones mantenidas hasta ahora, «no vemos grandes problemáticas porque son moléculas que no son capaces de autoreplicarse. Son cosas inertes en un tubito con agua», indica a EFE De la Fuente.
Aclara que esto no tiene nada que ver con la resurrección de organismos completos, pero reconoce que el debate sobre la desextinción molecular es necesario y debería involucrar a gobiernos y sociedad.
30 agosto 2024|Fuente: EFE |Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2024. Agencia Informativa Latinoamericana Prensa Latina S.A.|Noticia
ago
26
 La Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) autorizó hoy el uso de emergencia para las vacunas actualizadas contra la covid-19 en la próxima temporada de otoño e invierno.
La Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) autorizó hoy el uso de emergencia para las vacunas actualizadas contra la covid-19 en la próxima temporada de otoño e invierno.
El doctor Peter Marks, director del Centro de Evaluación e Investigación Biológica de la FDA, advirtió en un comunicado que «la vacunación sigue siendo la piedra angular de la prevención del Covid-19″.
«Dada la disminución de la inmunidad de la población por exposición previa al virus y por vacunación previa, alentamos encarecidamente a quienes son elegibles a considerar recibir una vacuna actualizada contra el covid-19 para brindar una mejor protección contra la variante que circula actualmente», subrayó el texto.
Según informó la agencia federal, los antídotos estarán dirigidos a la cepa KP.2, que es una rama de la variante ómicron del virus que provoca la enfermedad.
La FDA dijo que la decisión se aplica a las vacunas fabricadas por Pfizer-BioNTech y Moderna.
22 agosto 2024|Fuente: Prensa Latina |Tomado de |Noticia
ago
26
 Un algoritmo desarrollado por expertos del Instituto de Investigación en Biomedicina (IRB Barcelona) y del Centro de Regulación Genómica (CRG) puede predecir qué fármacos serán más efectivos para el tratamiento de enfermedades genéticas y del cáncer.
Un algoritmo desarrollado por expertos del Instituto de Investigación en Biomedicina (IRB Barcelona) y del Centro de Regulación Genómica (CRG) puede predecir qué fármacos serán más efectivos para el tratamiento de enfermedades genéticas y del cáncer.
El modelo computacional predictivo desarrollado es una herramienta de uso público llamada RTDetective y permite acelerar el diseño, desarrollo y eficacia de ensayos clínicos para una amplia variedad de trastornos causados por mutaciones en el ADN que provocan la síntesis de proteínas truncadas o incompletas.
Los hallazgos, publicados este jueves en Nature Genetics, marcan un paso importante en la personalización del tratamiento al emparejar a los pacientes con el fármaco más prometedor.
Las proteínas incompletas se originan cuando su síntesis se detiene repentinamente a causa de «mutaciones sin sentido» que actúan como una señal de stop o bloqueo.
En muchos casos, estas proteínas incompletas no pueden llevar a cabo su función y esto da lugar a diferentes trastornos.
Proteínas incompletas o inacabadas
De hecho, una de cada cinco enfermedades causadas por mutaciones en un único gen se relaciona con estas proteínas incompletas o inacabadas, incluyendo algunos tipos de fibrosis quística y la distrofia muscular de Duchenne.
Estas señales prematuras de stop también se dan en genes supresores de tumores, lo que provoca la inactivación de los mismos, favoreciendo el desarrollo del cáncer.
El estudio demuestra que, hasta la fecha, los ensayos clínicos de fármacos que funcionan leyendo a través de estas señales de stop probablemente han utilizado combinaciones de fármaco-paciente ineficaces.
Los investigadores desarrollaron un sistema experimental basado en líneas celulares humanas que les permitió medir la eficacia de ocho fármacos diferentes en 5 800 señales de stop prematuras causantes de enfermedades.
Así, descubrieron que un fármaco que funciona bien para superar una señal de stop prematura puede no ser efectivo para otra, incluso dentro del mismo gen, debido a la secuencia de ADN alrededor de la señal de stop.
Usaron el algoritmo para predecir la efectividad de fármacos
Los investigadores usaron el algoritmo para predecir la efectividad de diferentes fármacos para cada una de los 32,7 millones de posibles señales de stop que pueden generarse en el genoma humano.
Se predijo que al menos uno de los seis fármacos probados lograría un incremento del 1 % de lectura en el 87,3 % de todas las posibles señales de stop, y un incremento del 2 % en casi el 40 % de los casos.
Los resultados son prometedores porque porcentajes más altos de lectura generalmente se correlacionan con mejores resultados terapéuticos.
Los investigadores planean confirmar la funcionalidad de las proteínas producidas mediante fármacos, un paso clave para validar su aplicabilidad clínica.
El equipo también planea explorar otras estrategias que puedan usarse en combinación con estas terapias para aumentar la efectividad de los tratamientos, particularmente en el cáncer.
«Nuestro estudio no solo abre nuevas vías para el tratamiento de enfermedades genéticas hereditarias, sino también, y de manera importante, para el tratamiento de tumores», concluye Fran Supek, jefe del laboratorio de Ciencia de Datos del Genoma en el IRB Barcelona y profesor en el Centro de Investigación e Innovación Biotecnológica de la Universidad de Copenhague, quien ha coliderado el trabajo.
22 agosto 2024|Fuente: EFE |Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2024. Agencia Informativa Latinoamericana Prensa Latina S.A.|Noticia

