dic
11
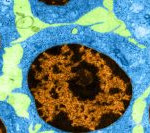 La célula leucémica corrompe la función normal de la cromatina, lo que bloquea la evolución hacia tipos celulares sanos y facilita el crecimiento tumoral, según ‘Nature Genetics’.
La célula leucémica corrompe la función normal de la cromatina, lo que bloquea la evolución hacia tipos celulares sanos y facilita el crecimiento tumoral, según ‘Nature Genetics’.
Un equipo internacional investigadores del CIMA Universidad de Navarra y de la Universidad de Cambridge ha descrito, por primera vez, los mecanismos de regulación genética que favorecen la evolución de la leucemia.
En este estudio multicéntrico, el más exhaustivo hasta el momento y que publicado en el último Nature Genetics, se han analizado las diferencias que existen en la generación de las células sanguíneas sanas frente a las células sanguíneas leucémicas.
En concreto, han caracterizado los mecanismos de regulación génica que utilizan las células para decidir cuándo y en qué medida un gen se activa o desactiva (expresión génica). Estudiar este proceso es muy importante ya que la regulación génica determina si la identidad que tomarán las células es de célula sana o de célula leucémica.
Usando tecnologías de última generación, los investigadores han desvelado que las células leucémicas corrompen mecanismos clave de regulación génica que determinan la identidad de las células sanas, lo cual bloquea su evolución hacia células sanas maduras y facilita el crecimiento del tumor.
Según el último informe de la Red Española de Registros de Cáncer (Redecan), en 2023 España contará con 6 411 nuevos casos de personas diagnosticadas de leucemia, el segundo cáncer de la sangre más prevalente. Así, este hallazgo abre la puerta al desarrollo de nuevos tratamientos para estos pacientes oncológicos.
Desregulación en el origen de la formación celular
La hematopoyesis comienza en las células madre hematopoyéticas, capaces de generar los distintos tipos de células sanguíneas (glóbulos blancos, glóbulos rojos y plaquetas). En concreto, es en la cromatina -la mezcla de ADN y proteínas que forman los cromosomas- donde se dan los procesos de regulación genética que dan lugar a la gran variedad de tipos celulares presentes en la sangre. En estos procesos intervienen dos grupos de proteínas llamados factores de la cromatina y factores de transcripción.
Los factores de transcripción marcan los genes específicos que se van a activar en cada tipo celular y los factores de la cromatina regulan la expresión de estos genes mediante cambios en la estructura bioquímica de la cromatina. De esta forma, se determina la identidad de las células sanguíneas. La desregulación de estos procesos desencadena distintos canceres sanguíneos, siendo la leucemia el segundo más frecuente.
Hasta el momento no estaba claro el papel que jugaban los factores de la cromatina en la determinación de la identidad celular. Sin embargo, este equipo de investigación ha demostrado que los factores de cromatina son un elemento crucial en la regulación de la identidad celular.
Para desmembrar esta función, han utilizado tecnologías de última generación de CRISPR y célula única.
A través del uso de tecnología que permite estudiar células individuales, hemos demostrado la complejidad de los procesos que regulan las células, revelando una gran diversidad en la función de factores de cromatina, así como otras funciones compartidas con los factores de transcripción», explican Julen Mendieta y Ainhoa Goñi, primeros autores de estudio e investigadores del Programa de Hemato-Oncología del CIMA, integrado en el Cancer Center Clínica Universidad de Navarra.
Diana ideal y específica
Estudiando los procesos que regulan la identidad celular en la leucemia, este equipo internacional ha revelado cómo las células leucémicas corrompen las funciones normales de los factores de cromatina para bloquear la evolución hacia tipos celulares sanos y facilitar el crecimiento tumoral.
En su análisis han observado que en esta alteración se formaron nuevos complejos de factores de transcripción y factores de cromatina exclusivos de las células leucémicas.
Según señala David Lara Astiaso, investigador del Departamento de Hematología de la Universidad de Cambridge y autor principal de estudio, como estos complejos son específicos de la leucemia y no son necesarios para la hematopoyesis normal, «son una diana ideal para una terapia que pueda desactivarlos sin causar ningún otro daño al paciente, a diferencia de tratamientos actuales como la quimioterapia, con altos niveles de toxicidad».
“Los fármacos epigenéticos están siendo muy útiles en ciertos linfomas y leucemias”
Una leucemia de ‘novo’ es un modelo de leucemogénesis
Nueva técnica para pronosticar la recaída en pacientes con leucemia mieloide aguda
Para el profesor Brian Huntly, director del Departamento de Hematología de la Universidad de Cambridge y codirector del estudio, identificar una nueva vía terapéutica potencial para la leucemia es especialmente importante. «Por ejemplo, en la leucemia mieloide aguda, que es la más común en adultos y muy agresiva, sólo el 15% de las personas diagnosticadas de esta enfermedad sobreviven más de cinco años».
En el desarrollo este estudio multicéntrico también ha colaborado investigadores la Universidad de Salzburgo (Austria) y de la compañía biotecnológica Relation Therapeutics (Reino Unido).
Varios de sus investigadores en España pertenecen al Centro de Investigación Biomédica en Red en Cáncer (CiberONC) y al Instituto de Investigación Sanitaria de Navarra (IdiSNA). El proyecto ha contado con la financiación de la Fundación Bancaria «la Caixa», de la Comisión Europea (Marie Skłodowska-Curie Actions) y del Cancer Research UK, Wellcome Trust, entre otras instituciones.
Ver más información: Lara-Astiaso D, Goñi-Salaverri A, Mendieta-Esteban J, Narayan N, Del Valle C, Gross T, et al. In vivo screening characterizes chromatin factor functions during normal and malignant hematopoiesis. Nat Genet [Internet].2023[citado 10 dic 2023];55, 1542:1554. https://doi.org/10.1038/s41588-023-01471-2
11 diciembre 2023 | Fuente: Diario Médico| Tomado de | Oncología
sep
21
 Investigadores norteamericanos han demostrado el potencial de los datos genéticos para informar la gestión de riesgos y la atención clínica de los pacientes con colesterol alto.
Investigadores norteamericanos han demostrado el potencial de los datos genéticos para informar la gestión de riesgos y la atención clínica de los pacientes con colesterol alto.
La hipercolesterolemia grave, entendida como una medición del colesterol LDL se asocia con un mayor riesgo de las arterias coronarias. Hay bastante evidencia de que niveles altos de LDL aumentan el riesgo de enfermedades cardíacas, ataques cardíacos y accidentes cerebrovasculares.
Para conocer el valor añadido de utilizar un enfoque genético para estratificar el riesgo de enfermedad cardíaca, se llevó a cabo un estudio por parte de los investigadores de Geisinger (EEUU) Matthew Oetjens, Alexander Berry, Laney Jones y Samuel Gidding.
Los resultados, publicados en ´Arteriosclerosis, Thrombosis and Vascular Biology´ «demuestra el potencial de los datos genéticos para informar la gestión de riesgos y la atención clínica de los pacientes con colesterol alto», indicó el Dr. Oetjens.
Para el trabajo, evaluaron datos genéticos de 11 738 participantes del Biobanco del Reino Unido que tenían niveles altos de LDL. El equipo observó una pequeña diferencia en la tasa de enfermedades cardíacas entre aquellos con los niveles más bajos y más altos de LDL.
Sin embargo, cuando los participantes fueron agrupados por la causa genética específica de su condición, hubo claras diferencias en las tasas de enfermedad. Por ejemplo, las personas con una variante genética única relacionada con el colesterol alto o con lipoproteína (a) elevada, una forma de colesterol LDL, tenían un riesgo significativamente mayor de enfermedad futura en comparación con el resto de los participantes del estudio.
Como parte del esfuerzo financiado por los Institutos Nacionales de Salud de Geisinger, RISK-FH, los investigadores tienen previsto investigar más a fondo la genética del riesgo de enfermedad cardíaca, utilizando biobancos que incluyen la Iniciativa de Salud Comunitaria MyCode de Geisinger, BioMe del Sistema de Salud Mt. Sinai y All of Us de los NIH.
Referencia
Berry ASF, Jones LK, Sijbrands EJ, Gidding SS, Oetjens MT. Subtyping Severe Hypercholesterolemia by Genetic Determinant to Stratify Risk of Coronary Artery Disease. Arterioscler Thromb Vasc Biol. 2023. doi: 10.1161/ATVBAHA.123.319341. Epub ahead of print. PMID: 3758913, .
https://pubmed.ncbi.nlm.nih.gov/37589137/
19/09/2023
Fuente: (IMMedico) Tomado Noticia- Cardiología
ago
23
 Se trata de la primera molécula interestelar con más de tres átomos de oxígeno. Un hallazgo que ayudará a comprender la química en el espacio que pudo ser clave en la aparición de la vida.
Se trata de la primera molécula interestelar con más de tres átomos de oxígeno. Un hallazgo que ayudará a comprender la química en el espacio que pudo ser clave en la aparición de la vida.
Un equipo internacional liderado por el Centro de Astrobiología español (CAB) descubrió ácido carbónico en el espacio, la primera molécula interestelar con más de tres átomos de oxígeno. Un hallazgo que ayudará a comprender la química interestelar, que pudo jugar un papel crucial para la aparición de la vida.
Material natal
Las principales teorías que explican el origen de la vida se basan en el desarrollo de una química prebiótica que tuvo lugar durante las primeras fases de la formación de nuestro planeta.
Se cree que una parte fundamental de los ingredientes prebióticos pudo llegar a una Tierra joven a bordo de cometas y meteoritos formados en la nebulosa donde nació el Sistema Solar.
Entender qué moléculas prebióticas están presentes en el medio interestelar, el material natal que formará nuevas estrellas y planetas, puede ser crucial para entender cómo pudo surgir la vida en nuestro planeta.
En los últimos años, se ha sugerido que puede haber ácido carbónico en diversos objetos del sistema solar, como las lunas heladas de Júpiter, el polo norte de Mercurio, o incluso en la superficie y atmósfera de Marte, pero hasta ahora no se habían encontrado evidencias que corroborasen su existencia extraterrestre.
Confirman presencia de ácido carbónico interestelar
El equipo multidisciplinar liderado por Miguel Sanz-Novo, en el Centro de Astrobiología (CAB, CSIC-INTA) ha demostrado la presencia de ácido carbónico en el espacio.
Esta molécula es la primera molécula interestelar que contiene tres átomos de oxígeno en su estructura y también el tercer ácido carboxílico detectado en el medio interestelar hasta el momento, después del ácido fórmico (identificado en el ISM en 1971) y el ácido acético (detectado en 1997). El equipo encontró la molécula en nuestra Vía Láctea.
«Nuestras observaciones nos han permitido saber que el ácido carbónico, que hasta ahora había permanecido invisible a nuestros ojos, es relativamente abundante en el espacio, lo que le convierte en una pieza esencial para entender la química interestelar del carbono y del oxígeno, dos de los elementos químicos fundamentales en cualquier proceso prebiótico», apunta Víctor M. Rivilla, investigador del CAB y coautor del artículo.
«Este resultado confirma que la senda que hemos escogido es la adecuada para buscar y detectar más moléculas que sospechamos fueron claves para la aparición de la vida en nuestro planeta», concluye.
Fuente: DW.COM
may
12
El 14 de abril de 2003 es una fecha importante para la historia de la ciencia. Aquel día de hace poco más de 20 años se anunció el fin del Proyecto Genoma Humano: la secuencia esencial de nuestro ADN había conseguido descifrarse después de muchos años de esfuerzo.
No obstante, ese ‘mapa’, que costó 3.000 millones de dólares y que comenzó a usarse como referencia del genoma humano, no estaba completo. Tenía lagunas en varias regiones genéticas y se basaba principalmente en el ADN de unos pocos individuos de origen europeo. Y aunque en estas dos décadas se han producido avances tecnológicos que han permitido ‘cartografiar’ esas lagunas -la secuencia completa se obtuvo en 2022- y abaratar el proceso, seguía faltando una referencia que fuera más global y diversa.
A partir de ahora, gracias a un consorcio internacional financiado por el Instituto de Investigación Nacional del Genoma Humano de EEUU, ese recurso -un pangenoma de referencia- estará disponible.
El nuevo ‘mapa’, que se presenta como un primer borrador, incluye la secuencia genética completa de 47 individuos de diferentes orígenes, lo que proporciona información detallada de 94 genomas debido a que cada individuo lleva en su ADN dos copias de genes ‘heredados’ de su padre y su madre. El objetivo del proyecto es seguir añadiendo datos al ‘mapa’, por lo que prevé que a mediados de 2024 incluya información genética de 350 personas de ascendencia étnica diversa.
«Hasta ahora, la referencia que usaba la comunidad científica estaba incompleta y carecía de diversidad», señaló en rueda de prensa Benedict Paten, director asociado del Instituto de Genómica Santa Cruz de la Universidad de California y uno de los líderes del proyecto. Este nuevo recurso, en cambio, proporciona una imagen más completa y permitirá realizar análisis más certeros a la hora de caracterizar la variabilidad genética de la población humana sea cual sea su origen, destacó.
De hecho, el nuevo pangenoma ya ha sacado a la luz más de 100 millones de nuevas bases -cada una de las letras que componen el genoma-, y ha destapado nuevos alelos en regiones estructuralmente complejas del genoma que hasta ahora no figuraban en el genoma de referencia. Los detalles de la investigación se publican en cuatro artículos en el último número de las revistas Nature y Nature Biotechnology.
Mediante técnicas computacionales de última generación, los investigadores han sido capaces de construir un recurso que, en lugar de ser único y lineal, como era hasta ahora la referencia GRCh38 que se utilizaba, aporta distintas versiones de una misma secuencia al mismo tiempo, lo que proporciona a los investigadores un mayor abanico de opciones para sus análisis. En el proyecto ha participado un equipo del Centro de Supercomputación de Barcelona liderado por Santiago Marco-Sola.
Qué supone un nuevo pangenoma para la investigación
«Hasta ahora nos hemos contentado con una sola secuencia del genoma que en su día se decidió arbitrariamente que era la secuencia referencia, formada por trozos de secuencia de un puñado de personas de ascendencia principalmente europea. Y si bien esto ha tenido una gran utilidad, también tiene muchas limitaciones», señala Jorge Ferrer, investigador del Centro de Regulación Genómica de Barcelona (CRG). «Por ejemplo, aunque resulte sorprendente, a cada uno de nosotros nos pueden faltar o sobrar unos cuantos trozos muy grandes del genoma. Si el pedazo de genoma escogido para ser la referencia es de alguien que no tiene ese trozo (o lo tiene suficientemente alterado), el mapa de referencia que utilizamos actualmente no serviría para una persona que tiene una mutación que afecta esa parte», aclara. Para complicar aún más las cosas, continúa, «el genoma puede variar enormemente en diferentes partes del mundo. Y si el mapa de referencia está hecho con variantes europeas, tiene menor utilidad para interpretar el genoma de una persona de Camerún o China».
El trabajo actual, apunta Ferrer, «es el primer paso para resolver estos problemas». «Han creado un sistema complejo que permite cotejar la secuencia genómica de una persona con todas estas posibles secuencias humanas, en lugar de con una sola secuencia y el consorcio tiene planes de desarrollar esta estrategia con la secuencia de muchos más individuos».
Para José Manuel Castro Tubío, líder del Grupo de Investigación de Genomas y Enfermedad del Centro de Investigación en Medicina Molecular y Enfermedades Crónicas (CIMUS) de Santiago, este nuevo recurso va a ayudar, en primer lugar, a «conocer mejor nuestra identidad, a conocer qué es lo que nos hace diferentes genéticamente a unos de otros». Y el hecho de «conocer lo que nos hace diferentes, qué secuencias de material genético nos hacen diferentes, nos va a permitir saber cosas acerca de nuestra evolución y nos va a permitir también saber cosas sobre las enfermedades genéticas que nos afectan».
«La variabilidad genética va asociada a rasgos biológicos y también a la predisposición de desarrollar enfermedades», explica. «Estos nuevos genomas que ahora se publican van a permitir descubrir muchas variantes que todavía no sabemos a qué se asocian».
Si bien la nueva referencia presentada todavía es un primer borrador y solo representa a un número todavía pequeño de individuos, contiene información que va a ser muy útil para avanzar en la investigación biomédica, concluye el investigador.
Mayo 10/2023 (Diario Médico) – Tomado de Genética – Investigación Copyright Junio 2018 Unidad Editorial Revistas, S.L.U. Todos los derechos reservados
feb
16
El director de la Organización Mundial de la Salud (OMS) se comprometió recientemente a hacer todo lo que sea posible para tener una ‘respuesta’ sobre el origen del covid-19. Read more
jun
11
China rechazó y consideró una mentira con tintes políticos, la teoría sobre la supuesta fuga del coronavirus SARS-CoV-2 de un laboratorio de Wuhan, en 2019, tras indicar que está abierta a las investigaciones sin manipulación. Read more


