mar
25
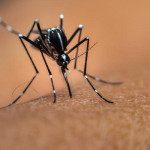 Al comparar datos referentes a la respuesta inmunitaria resultante de una infección natural causada por el dengue con los de la activación inmunológica generada por las vacunas contra la enfermedad, investigadores de la Universidad de São Paulo (USP), en Brasil, identificaron marcadores moleculares que podrán servir en el futuro para desarrollar nuevas vacunas y tratamientos contra este virus.
Al comparar datos referentes a la respuesta inmunitaria resultante de una infección natural causada por el dengue con los de la activación inmunológica generada por las vacunas contra la enfermedad, investigadores de la Universidad de São Paulo (USP), en Brasil, identificaron marcadores moleculares que podrán servir en el futuro para desarrollar nuevas vacunas y tratamientos contra este virus.
Este trabajo también reiteró la eficacia de la respuesta inmune inducida por dos inmunógenos disponibles en el mercado: Q-Denga, desarrollado por el laboratorio japonés Takeda Pharma y actualmente distribuido por el Sistema Único de Salud de Brasil (el SUS, la red nacional de salud pública del país), y Dengvaxia, del laboratorio francés Sanofi-Pasteur. En ambos se aplica la tecnología de virus atenuado.
Este estudio, publicado en la revista Frontiers in Immunology, es el primero en el cual se identifican firmas inmunológicas del dengue mediante la aplicación de un abordaje denominado vacunología sistémica, un campo de investigación que apunta a descifrar un cuadro global de las respuestas inmunológicas a la vacunación.
Efectuamos un abordaje sistémico e identificamos diversas similitudes entre la respuesta inducida por la vacuna y la de una infección natural provocada por el virus. Por supuesto que, en el caso de los inmunógenos, los mismos promovieron la respuesta inmunitaria sin causar el daño que causa el dengue. En el estudio también logramos caracterizar algunas vías [de señalización entre células de defensa]. Y la vía del interferón [mediada por esta proteína antiviral producida por los leucocitos y por fibroblastos] se mostró central, con diversos genes importantes que surgen como nuevos biomarcadores de la enfermedad”, comenta Otávio Cabral-Marques, docente de la Facultad de Medicina de la USP (FM-USP) y coordinador de la investigación.
En el referido trabajo, los investigadores analizaron 955 muestras de transcriptomas (conjuntos completos de moléculas de ARN expresadas) de pacientes infectados por el mosquito del dengue y de participantes en ensayos clínicos de compuestos inmunizantes contra la enfermedad. Los datos se obtuvieron en bancos públicos. De este modo, se identificaron 237 genes expresados diferentemente tanto en los casos de infección natural como en los de respuesta a las vacunas.
Con base en 20 de esos genes en común, logramos crear un panel para distinguir la gravedad de la enfermedad, particularmente en la fase aguda tardía. Mediante la aplicación de técnicas de aprendizaje automático, también fue posible clasificar diez predictores [firmas inmunológicas] de gravedad de la enfermedad en los casos de infección natural que son cruciales para la respuesta inmunitaria antiviral”, explica Desirée Rodrigues Plaça, autora principal del estudio y becaria doctoral de la FAPESP.
Aparte de ser causada por cuatro serotipos virales (DENV-1, 2, 3 y 4), el dengue lleva la impronta de diferentes fases clínicas. Cuando no es asintomática, la enfermedad exhibe una fase aguda inicial, una fase aguda tardía y una fase convaleciente.
Existen muchas diferencias, pero también logramos identificar muchas similitudes entre la respuesta inmune inducida por las vacunas y aquella que la infección natural provoca. Al tamizar el conjunto de datos, identificamos 20 genes comunes a esos dos procesos, que se expresan de manera análoga. Son los responsables de enriquecer la vía protectora del sistema inmunitario, sobre todo la vía inmunológica del interferón tipo 1 y 2”, dice Rodrigues Plaça.
El interferón es una citoquina cuyo rol principal consiste en inhibir la replicación viral. Como provoca una reacción en cadena que afecta a varias moléculas, el estudio demostró que, si bien las vías antivirales del interferón son responsables de una defensa temprana (actúan en la primera línea), a través de sus complejas funciones biológicas la proteína prepara el terreno para el desarrollo de una inmunidad adaptativa robusta y duradera.
La vía del interferón es sumamente importante en la activación de la respuesta adaptativa. Formada por las células T y B [dos tipos de linfocitos], esta respuesta adaptativa genera la protección permanente [el objetivo de la vacuna]. Por ende, resulta de suma importancia entender qué genes asociados a esta vía serían determinantes para producir una respuesta adaptativa más protectora”, explica Rodrigues Plaça.
Con la información obtenida en el estudio, los investigadores lograron determinar estrategias terapéuticas que pueden bloquear, activar o inducir la actuación de genes implicados en el proceso de la respuesta inmunitaria, lo que abre la posibilidad de investigar nuevos blancos terapéuticos para la enfermedad.
En el trabajo, los científicos identificaron los principales genes
(OAS2, ISG15, AIM2, OAS1, SIGLEC1, IFI6, IFI44L, IFIH1 e IFI44) implicados en el entramado de aspectos importantes de la respuesta inmune adaptativa. De esta forma, mientras que algunos genes (OAS2 y OAS) exhiben funciones antivirales, otros (como ISG15) restringen la replicación del virus.
Los investigadores descubrieron también que en este proceso existe un gen (AIM2) responsable de activar el inflamasoma un complejo proteico existente en el interior de las células de defensa que cuando se activa produce moléculas que le avisan al sistema inmunitario acerca de la necesidad de enviar refuerzos al lugar de la infección, lo que pone en marcha respuestas proinflamatorias cruciales para lograr una defensa antivírica eficaz.
Le compete al gen SIGLEC1 participar en las interacciones de las células inmunitarias facilitando la presentación de antígenos y el reconocimiento inmunológico adaptativo. Inducidos por el interferón, tres genes específicos (IFI6, IFI44L e IFI44) están asociados a la modulación de la apoptosis (la muerte celular programada) y a la defensa antiviral. En tanto, el gen IFIH1 opera como un sensor importante, que modula la respuesta adaptativa a los virus de ARN, tal como es el caso del DENV. Otros genes (IFIT5 y HERC5) exhiben un papel importante en la inhibición de la replicación viral mediada por el interferón.
El conjunto de genes relevantes en todo este proceso remarca cuán intrincada es la ligazón entre las vías antivirales iniciales y los procesos inmunitarios adaptativos subsiguientes. Es como si las dos puntas de una historia se uniesen”, comenta Cabral-Marques.
Ver artículo: Rodriguez Placa D, Fonseca DL, Marquez AH, Zaki Pour S, Nakanishi Usuda J, Crispim Baiocchi G, et al. Immunological signatures unveiled by integrative systems vaccinology characterization of dengue vaccination trials and natural infection. Front Immunol[Internet]. 2024[citado 22 mar 2024]; 15. https://doi.org/10.3389/fimmu.2024.1282754
22 marzo 2024| Fuente: Dicyt| Tomado de | Noticias| Salud
mar
18
 El científico de la Facultad de Medicina de la Universidad de Málaga, Jose Luis Royo ha liderado un estudio que resume ocho años de investigación y a un equipo de un centenar de especialistas multidisciplinares, a través del cual se ha identificado una mutación que altera la progresión de la enfermedad de Alzheimer. Este trabajo publicado en la revista Journal of Alzheimers Disease’, ha caracterizado una variante genética en el gen SIRPB1, que afecta el modo en el que el sistema inmune lucha contra los depósitos de beta-amiloide, causantes de esta patología.
El científico de la Facultad de Medicina de la Universidad de Málaga, Jose Luis Royo ha liderado un estudio que resume ocho años de investigación y a un equipo de un centenar de especialistas multidisciplinares, a través del cual se ha identificado una mutación que altera la progresión de la enfermedad de Alzheimer. Este trabajo publicado en la revista Journal of Alzheimers Disease’, ha caracterizado una variante genética en el gen SIRPB1, que afecta el modo en el que el sistema inmune lucha contra los depósitos de beta-amiloide, causantes de esta patología.
Para su desarrollo, se llevó a cabo un estudio longitudinal a partir de muestras epidemiológicas de más de 1.300 pacientes de Málaga y Barcelona, que evidenció el papel de esta mutación en el declive cognitivo de los afectados a lo largo de la enfermedad. No obstante, se constató que no representa un factor de riesgo en sí para padecer alzhéimer.
“Todos los seres humanos tenemos los mismos genes, pero de cada uno existen variantes que nos hacen diferentes. Distintas apariencias exteriores y, también, diferentes fisiologías y metabolismos. Son esas diferencias genéticas las que condicionan nuestro metabolismo”, explica el profesor del Departamento de Especialidades Quirúrgicas, Bioquímica e Inmunología Jose Luis Royo.
El científico de la UMA señala que cuando se ha analizado la presencia de esta variante del gen SIRPB1 se ha descubierto que un 30 por ciento de la población general tiene una copia de mutación y un 4 por ciento de la población tiene las dos copias afectadas. “Por lo tanto hablamos de una variante que está muy presente en la población general”, asegura.
Efecto dual
La investigación, en la que participan científicos de más de 50 entidades diferentes, muestra que la variante mutante cambia la estructura de la proteína y como esto altera el comportamiento de las células inmunes cerebrales, denominadas microglía.
“Cuando hemos estudiado el papel que tiene en el transcurso de la enfermedad, hemos visto que tiene un efecto dual dependiendo de la fase de la misma: al principio, en pacientes de deterioro cognitivo leve, la mutación aumenta la tasa de conversión a alzhéimer, por lo que tiene un efecto perjudicial en las fases iniciales de la enfermedad; sin embargo, cuando la demencia se establece como tal, aquellos portadores de la mutación muestran un deterioro cognitivo más lento, lo que sugiere un efecto beneficioso entre estos pacientes”, aclara al investigador de la UMA.
Royo precisa que, por tanto, a lo que afecta la mutación es a la reacción del sistema inmune a los depósitos de beta-amiloide, probablemente al proceso de neuroinflamación, que es una respuesta fisiológica en los estadios iniciales, pero perjudicial si se hace crónico cuando avanza la enfermedad.
Así, este efecto dual de la variante genética, según afirma, sugiere que esta ruta de señalización celular debería inhibirse durante los estadios iniciales de la enfermedad, pero en pacientes en estadios más avanzados se debería estimular, para mimetizar, de forma química, el efecto que genera la mutación. “Este hallazgo abre la puerta a una nueva diana terapéutica bioquímica que, en el futuro, se podría plasmar en sintetizar con fármaco”, concluye.
Actualmente, se sigue trabajando para caracterizar a un mayor nivel de complejidad el efecto de esta mutación y se ha diseñado un sistema para buscar modificadores de esta ruta de señalización celular.
Ver artículo: García Alberca JM, Rojas I de, Sanchez Mejias E, Garrido Martín D, Gonzales Palma L, Jimenez S, et al. An Insertion Within SIRPβ1 Shows a Dual Effect Over Alzheimer’s Disease Cognitive Decline Altering the Microglial Response. J Alzheimers Dis[Internet] . 2024[citado 18 mar 2024]. doi: 10.3233/JAD-231150. https://pubmed.ncbi.nlm.nih.gov/38427484/
18 marzo 2024|Fuente: EureKalert| tomado de| Comunicado prensa
mar
12
 Un reciente estudio publicado en JAMA Pediatrics concluye que la actividad de cuatro genes puede predecir la gravedad de la apendicitis en pacientes pediátricos. La apendicitis, que es la inflamación del apéndice, es la urgencia quirúrgica más común en niños.
Un reciente estudio publicado en JAMA Pediatrics concluye que la actividad de cuatro genes puede predecir la gravedad de la apendicitis en pacientes pediátricos. La apendicitis, que es la inflamación del apéndice, es la urgencia quirúrgica más común en niños.
Producida generalmente debido a un bloqueo del apéndice que deriva en una infección, la inflamación del apéndice se manifiesta a través del dolor abdominal. En la mayoría de los casos, el diagnóstico es sencillo, aunque la falta de especificidad en la presentación clínica puede dificultar el proceso. El problema radica en estimar el grado de gravedad.
El tratamiento de la apendicitis suele consistir en una intervención quirúrgica para extirpar el apéndice y evitar su perforación lo que puede extender la infección por el organismo. Además, se ha planteado que en ciertos casos leves el tratamiento con antibióticos puede ser una opción a valorar.
En este contexto, diferenciar entre apendicitis simple y apendicitis aguda con perforación en niños puede mejorar el diagnóstico y contribuir a determinar mejor el tratamiento. Con este objetivo, un reciente estudio publicado en JAMA Pediatrics plantea una prueba molecular para complementar los métodos estándar de determinación de la gravedad de la apendicitis en niños: un análisis de expresión de cuatro genes que permite diferenciar entre las formas más graves y leves de la apendicitis.
Expresión génica relacionada con la gravedad de la apendicitis
Para identificar biomarcadores relacionados con la progresión o gravedad de la apendicitis los investigadores analizaron la expresión génica en muestras de sangre de 71 niños entre 5 y 17 años que ingresaron en el Hospital de Niños de Alberta (Canadá) con dolor abdominal y sospecha de apendicitis. De estos pacientes, 35 tuvieron apendicitis simple, 14 mostraron perforación en apéndice y en 22 no se confirmó apendicitis.
El equipo observó cambios significativos en la expresión génica de los niños del grupo con apendicitis perforada, relacionados con la función inmunitaria y más concretamente con una disminución en la respuesta inmunitaria e inflamatoria. En estos pacientes, el perfil de expresión mostró similitudes con el reportado en pacientes con sepsis, lo que es consistente con los procesos que se espera que ocurran en un escenario de perforación del apéndice.
Además, los investigadores identificaron una firma de expresión de cuatro genes que permitía diferenciar la apendicitis perforada de la apendicitis simple, con una precisión del 85.7%. Los cuatro genes son PLBD1, S100A12, S100A8 y ANXA3 y en todo ellos la expresión está aumentada en pacientes con apéndice perforado. S100A12 y S100A8 codifican proteínas de la familia S100 y son conocidos moduladores del sistema inmunitario. De hecho, S100A12 ya se ha sugerido como biomarcador de diversas condiciones inflamatorias y digestivas. ANXA3, que pertenece a la familia de las anexinas, también se ha propuesto como biomarcador de sepsis en pacientes pediátricos.
Biomarcadores genéticos para una mejor predicción del riesgo en apendicitis
La identificación de biomarcadores genéticos capaces de predecir la severidad de la apendicitis en niños puede tener implicaciones en el manejo clínico de esta condición.
En primer lugar, ofrece una herramienta diagnóstica poco invasiva (basada en un análisis de sangre) que, junto a métodos tradicionales, puede favorecer una estratificación más precisa del riesgo y, por tanto, un manejo de los pacientes mejor adaptado a cada situación.
Por ejemplo, en los casos donde se estime mayor gravedad se puede proceder rápidamente a la intervención quirúrgica para minimizar el riesgo de complicaciones graves. “El desarrollo de estrategias de diagnóstico precoz y tratamiento de la apendicitis con perforación pediátrica debe basarse en la desregulación inmunitaria subyacente y en las similitudes con la sepsis”, señalan los investigadores en el trabajo. Además, el estudio proporciona una estimación de los procesos inmunitarios que pueden estar implicados en la apendicitis, especialmente cuando se trata de casos más graves.
Artículo científico: Dhillon BK, Kortbeek S, Baghela A, Brindle M, Martin DA, Jenne CN, Vogel HJ, Lee AHY, Thompson GC, Hancock REW. Gene Expression Profiling in Pediatric Appendicitis. JAMA Pediatr. 2024 Feb 19:e236721. doi: http://dx.doi.org/10.1001/jamapediatrics.2023.6721.
07 marzo2024|Fuente:Genotipia|Tomado de | Noticias
ene
25
 El glaucoma entre las personas afroamericanas parece estar relacionado con tres variantes genéticas, sugiere un estudio reciente
El glaucoma entre las personas afroamericanas parece estar relacionado con tres variantes genéticas, sugiere un estudio reciente
En las personas afroamericanas, una puntuación de riesgo con estas tres variantes superó a una puntuación de riesgo similar basada en datos de personas blancas
Las personas afroamericanas tienen cinco veces más probabilidades de desarrollar glaucoma y 15 veces más probabilidades de quedar ciegas por el padecimiento
Las personas afroamericanas tienen cinco veces más probabilidades que los demás de desarrollar glaucoma, y hasta 15 veces más probabilidades de quedar ciegas por la enfermedad ocular degenerativa.
Ahora, un nuevo estudio informa que la genética parece ser al menos un factor que contribuye a este aumento del riesgo.
Los investigadores han identificado tres variantes genéticas que podrían estar alimentando el mayor riesgo de glaucoma de las personas afroamericanas, según los hallazgos, que se publicaron en la edición del 18 de enero de la revista Cell.
«Nuestro trabajo es un paso importante hacia la definición de subgrupos de glaucoma, proporcionando la capacidad de una evaluación temprana y el descubrimiento de vías dirigidas para intervenciones terapéuticas personalizadas», señaló la coautora del estudio, Rebecca Salowe, directora de proyectos de investigación del Centro de Genética de Enfermedades Complejas de la Universidad de Pensilvania, en Filadelfia.
El glaucoma ocurre cuando la presión del líquido comienza a aumentar dentro del ojo de una persona, dañando gradualmente el nervio óptico.
En el estudio, los investigadores analizaron la genética de más de 11,200 personas de ascendencia africana.
Descubrieron dos variantes genéticas relacionadas con el glaucoma primario de ángulo abierto, que es la forma más común de la enfermedad.
El glaucoma de ángulo abierto afecta hasta a nueve de cada 10 estadounidenses que tienen glaucoma, según la Clínica Cleveland. Ocurre cuando se acumula resistencia en los canales que normalmente drenan el exceso de líquido del ojo.
También encontraron una tercera variante asociada con la relación copa-disco, que es una medida de la gravedad del glaucoma basada en la forma del ojo de una persona.
Utilizando las variantes detectadas en este estudio, los investigadores desarrollaron una puntuación de riesgo genético para el glaucoma en pacientes afroamericanos que superó una puntuación de riesgo similar basada en datos de personas de ascendencia europea.
Esta puntuación podría ayudar a los pacientes con un riesgo elevado de glaucoma a obtener una detección y un tratamiento tempranos para la enfermedad, antes de que los deje ciegos.
«El glaucoma es una enfermedad altamente familiar, por lo que la pérdida prematura e irreversible de la visión puede afectar a varios miembros de la familia, lo que contribuye tanto a resultados adversos para la salud como para la economía», dijo Salowe. «Dado que los tratamientos actuales para el glaucoma tienen un éxito limitado, existe una necesidad urgente de grandes estudios genéticos para identificar nuevos objetivos para la detección y la intervención terapéutica en individuos de ascendencia africana».
Hasta ahora, solo un 2 por ciento de los estudios que vinculan la genética con enfermedades específicas han involucrado a personas cuya ascendencia está vinculada a África, anotaron los investigadores.
«Este trabajo resalta el rol esencial de la diversidad en la investigación genética», señaló la investigadora principal, Shefali Setia Verma, profesora asistente de patología y medicina de laboratorio de la Universidad de Pensilvania.
Referencia: Verma SS, Gudiseva HV, Chaval VR, Hauser MA, Ritchie MD, O’Brien JM, et al. A multi-cohort genome-wide association study in African ancestry individuals reveals risk loci for primary open-angle glaucoma. Cell [Internet].2024[citado 23 ene 2024];187(2):464-480. DOI:https://doi.org/10.1016/j.cell.2023.12.006
23 enero 2024│ Fuente: HealthDay │ Tomado de Noticias de Salud
ene
17
 Un equipo de investigadores del español Centro Nacional de Investigaciones Oncológicas (CNIO) ha descubierto 103 genes con alteraciones, que causan enfermedades hereditarias y que, a la vez, aumentan el riesgo de desarrollar cáncer.
Un equipo de investigadores del español Centro Nacional de Investigaciones Oncológicas (CNIO) ha descubierto 103 genes con alteraciones, que causan enfermedades hereditarias y que, a la vez, aumentan el riesgo de desarrollar cáncer.
Los detalles de la investigación, liderada por la jefa del grupo de Genómica Computacional del Cáncer en el CNIO, Solip Park, y hecha en colaboración con centros de Seúl (Corea del Sur), se ha publicado en Genome Medicine.
La probabilidad de desarrollar cáncer es mayor entre los que tienen variantes alteradas de los genes de predisposición al cáncer (CPG), pero estos solo explican uno de cada diez casos de cáncer, los demás podrían estar relacionados por mutaciones desconocidas. Por ahora solo se conocen un centenar de CPG. Encontrar otras variantes alteradas ayudaría a detectar el cáncer y a desarrollar tratamientos. Para descubrirlos, Park acotó la búsqueda a las personas portadoras de genes que, cuando están alterados, dan lugar a una enfermedad hereditaria.
Se trata de enfermedades monogénicas (se producen por la alteración de un solo gen), como la distrofia muscular o la enfermedad de Gaucher por la que se acumulan grasas en diversas células–. Así, junto a varias instituciones coreanas, descubrieron 103 genes en los que las alteraciones que causan enfermedades monogenéticas a menudo coexisten con otras alteraciones que predisponen al cáncer.
El estudio verificó que las personas con mutaciones de enfermedades monogenéticas hereditarias en esos 103 genes también presentaban mayor cantidad de mutaciones implicadas en cáncer que el grupo de control, de personas sanas.
Algunas de estas mutaciones se asocian a tipos específicos de cáncer, como carcinoma de células renales, linfoma no Hodgkin de células B, adenocarcinoma de mama y meduloblastoma; otras, con la propensión al cáncer en general. El equipo analizó también cómo las variantes defectuosas de esos genes promueven la progresión de los tumores y causan otras enfermedades. Uno de ellos es el gen PAH, que fue seleccionado porque presentaba la mayor cantidad de variantes susceptibles de dar lugar a varios tipos de cáncer. El equipo encontró que este gen está relacionado con el carcinoma de células escamosas de pulmón, con tumores del tejido hepático, con otras enfermedades y con un retraso en el crecimiento.
Ver artículo completo: Song S, Koh Y, Kim S, Mi Lee S, Uk Kim H, Min Ko J, et al. Systematic analysis of Mendelian disease-associated gene variants reveals new classes of cancer-predisposing genes. Genome Medicine [Internet]. 2023; 07. https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-023-01252-w
Enero 17 /2024(EFE) – Tomado de la Selección Temática sobre Medicina de Prensa Latina. Copyright 2019. Agencia Informativa Latinoamericana Prensa Latina S.A
dic
6
 La mayoría de las oncoproteínas humanas pertenecen a una clase de proteínas llamadas “factores de transcripción”, pero hasta ahora no se había conseguido diseñar medicamentos para bloquear o amplificar su efecto.
La mayoría de las oncoproteínas humanas pertenecen a una clase de proteínas llamadas “factores de transcripción”, pero hasta ahora no se había conseguido diseñar medicamentos para bloquear o amplificar su efecto.
Un equipo internacional de investigadores, liderado por el IRB Barcelona, ha descubierto un nuevo enfoque para dirigirse al receptor de andrógenos, un factor de transcripción clave en el cáncer de próstata, basado en su propensión a formar condensados.
Los resultados descritos en esta publicación sentaron las bases para la creación de Nuage Therapeutics, spin-off de IRB Barcelona e ICREA.
Los resultados se publican en Nature Structural & Molecular Biology.
Barcelona, 4 de diciembre de 2023 – Los factores de transcripción son proteínas reguladoras que se unen al ADN, activan o desactivan genes y controlan la velocidad a la que se transcribe el ADN en ARN mensajero, necesario para la síntesis de proteínas. Debido a su papel central en el control de la formación de nuevas proteínas, muchas enfermedades pueden atribuirse a factores de transcripción desregulados.
Inhibir su actividad, especialmente en el cáncer, ofrece un potencial terapéutico, pero muchos factores de transcripción esconden un as bajo la manga. Sus dominios de activación son intrínsecamente desordenados, lo que significa que carecen de una estructura tridimensional clara. La falta de una estructura 3D estable hace prácticamente imposible diseñar medicamentos que se unan a los dominios de activación.
Un equipo liderado por los investigadores del IRB Barcelona, Dr. Xavier Salvatella (Profesor de Investigación ICREA) y Dr. Antoni Riera (Profesor de la Universidad de Barcelona), y por el Dr. Denes Hnisz del Instituto Max Planck de Genética Molecular, y la Dra. Marianne D. Sadar del BC Cancer (Universidad de British Columbia, Canadá) se centró en la tendencia de las proteínas intrínsecamente desordenadas a formar condensados moleculares. Descubrieron que los mecanismos involucrados en la condensación podrían ser utilizados para inhibir la actividad del receptor de andrógenos en el cáncer de próstata.
«La lógica que hemos seguido para optimizar un inhibidor del receptor de andrógenos podría ser explotada para inhibir otros factores de transcripción, lo que abre nuevas posibilidades para abordar necesidades médicas no cubiertas», explica el Dr. Salvatella, jefe del laboratorio de Biofísica Molecular del IRB Barcelona.
Los condensados, un nuevo enfoque para dirigirse a los factores de transcripción
En el microscopio, los condensados moleculares parecen manchas de proteína flotando en agua. Los condensados se forman en un proceso llamado “separación de fases líquido-líquido”, similar a cómo las gotas de aceite se fusionan cuando se mezclan en agua.
«Habíamos observado anteriormente que el receptor de andrógenos forma condensados moleculares cuando se agrega incluso una pequeña cantidad de una molécula activadora, como la testosterona, a las células», explica el Dr. Shaon Basu, actualmente biólogo computacional en Charité en Berlín, y uno de los primeros autores del estudio junto con la Dra. Paula Martínez-Cristóbal del IRB Barcelona.
Los científicos plantearon la hipótesis de que podría haber una conexión entre la activación del receptor de andrógenos y su propensión a formar condensados. En el laboratorio del Dr. Salvatella, se utilizaron técnicas de resonancia magnética nuclear para identificar varios fragmentos cortos dentro del dominio de activación intrínsecamente desordenado que son esenciales para la separación de fases.
Además, estos fragmentos cortos resultaron ser también necesarios para la función de activación génica del receptor. «Descubrimos secuencias cortas en el dominio de activación que tienden a estar desordenadas cuando la proteína es soluble, y sorprendentemente, estas regiones parecen formar hélices más estables cuando la proteína se concentra en condensados», añade el Dr. Hnisz. Las hélices cortas crean puntos de unión transitoria que pueden ser el objetivo de inhibidores cuando, el receptor está en forma de condensados.
Mejora de los compuestos para el tratamiento del cáncer de próstata
Trabajando con los laboratorios del Dr. Antoni Riera y la Dra. Marianne Sadar, el equipo mejoró un inhibidor experimental de molécula pequeña para adaptarse casi perfectamente a estos puntos transitorios. Luego, probaron en modelos celulares y de ratones si estos cambios aumentarían la eficacia en una forma agresiva y en una etapa avanzada de cáncer de próstata.
«Modificamos las características químicas del compuesto para que coincidan con las características de la condensación del receptor de andrógenos, lo que resultó en un aumento diez veces mayor en la potencia de la molécula en el cáncer de próstata resistente a la castración», explica la Dra. Paula Martínez-Cristóbal, también primera autora del estudio. «Esto es realmente importante, porque el cáncer de próstata resistente a la castración es extremadamente agresivo y es resistente a los tratamientos actuales», añade.
Sin embargo, los autores coinciden en que se necesita más investigación antes de que estos hallazgos se traduzcan en terapias nuevas y seguras. El equipo espera que los mecanismos básicos que han descubierto puedan aplicarse a otros factores de transcripción, abriendo la puerta a dirigirse a estas importantes moléculas en muchas enfermedades. «Creemos que la idea de que ciertas secuencias dentro de los dominios de proteínas intrínsecamente desordenadas adopten una estructura transitoria estable en forma de condensados es probablemente generalizable a otros factores de transcripción», concluye el Dr. Hnisz.
La empresa biotecnológica Nuage Therapeutics
Fundada por el Dr. Xavier Salvatella, el Dr. Mateusz Biesaga, Denes Hnisz y la Dra. Judit Anido, la empresa biotecnológica Nuage Therapeutics desarrolla ensayos de cribado de fármacos dirigidos a proteínas intrínsecamente desordenadas que experimentan condensación biomolecular, proporcionando así nuevos tratamientos para enfermedades que actualmente se consideran difíciles de tratar.
Los hallazgos publicados en Nature Structural & Molecular Biology sentaron las bases para la fundación de esta empresa en septiembre de 2021. El potencial de su ciencia llevó a una ronda de financiación inicial de 12 millones de euros en junio de 2023.
Ver más información: Basu S, Martínez Cristóbal P, Frigolé Vivas M, Pesarrodona M, Lewis M, Szulc E, et al. Rational optimization of a transcription factor activation domain inhibitor. Nature Structural & Molecular Biology [Internet].2023[citado 4 dic 2023]; DOI 10.1038/s41594-023-01159-5
6 diciembre 2023|Fuente: EurekAlert| Tomado de | Comunicado de Prensa

